Версія даної теми для друку
Натисніть сюди для перегляду даної теми у оригінальному форматі
Розподілені обчислення в Україні _ Rosetta@Home _ Rosetta@home
Автор: Dimych May 29 2007, 12:12

Проект "Rosetta@home"
----------------------------------------------------------------------------------------------------------
http://boinc.bakerlab.org/rosetta/
http://boinc.bakerlab.org/rosetta/team_members.php?teamid=328&sort_by=total_credit&offset=0
ТОП-20 участников:
----------------------------------------------------------------------------------------------------------
Дата основания команды - 12.04.2006 Капитан - uNiUs
----------------------------------------------------------------------------------------------------------
Для присоединения к команде Украины:
1. Загрузите http://boinc.berkeley.edu/download.php (Если его у Вас еще нет!)
2. Перейдите в "расширенный вид"
3. Выберите сервис ---> добавить проект
4. Введите адрес проекта http://boinc.bakerlab.org/rosetta
5. Введите свои регистрационные данные.
6. Найдите нашу команду. Она называется Ukraine и адрес ее http://boinc.bakerlab.org/rosetta/team_members.php?teamid=328&sort_by=total_credit&offset=0 вы могли видеть выше.
7. Если есть доступные для загрузки задания Вы их получите и начнете расчеты.
----------------------------------------------------------------------------------------------------------
http://boinc.berkeley.edu/dev/forum_thread.php?id=2518
----------------------------------------------------------------------------------------------------------
Полезная информация:
Для идентификации пользователя в BOINC могут служить 2 вещи:
1) пара e-mail/пароль
2) межпроектный идентификационный ID (Cross-project ID) - 32значное шестнадцатиричное число.
Если Вы пожелаете подключится ещё и к другому BOINC-проекту, то помните: чтобы не плодить новых аккаунтов при подключении к новому проекту или команде, нужно обязательно везде регистрироваться с одним и тем же e-mail/паролем либо CPID. если при регистрации в проекте указать другие e-mail или пароль, BOINC создаст новый аккаунт с тем же именем!
----------------------------------------------------------------------------------------------------------
Если у вас есть желание помочь команде, посчитать на командный аккаунт, можно использовать так называемый "слабый ключ аккаунта" (weak account key) который позволяет коннектиться к проекту не вводя пароли. Все очки будут присваиваться командному юзеру http://boinc.bakerlab.org/rosetta/show_user.php?userid=418972
Чтобы подключиться:
0) установите BOINC
1) создайте файл account_boinc.bakerlab.org_rosetta.xml в папке \data\
2) в файл поместите текст
<master_url>http://boinc.bakerlab.org/rosetta/</master_url>
<authenticator>46ed7286c5974c66fe43ee6b4828413a</authenticator>
<project_name>rosetta@home</project_name>
</account>
3) перезагрузите BOINC
----------------------------------------------------------------------------------------------------------
Описание проекта:
Цель проекта - решение одной из самых больших проблем в молекулярной биологии - вычисление 3-х мерной структуры белков из их аминокислотных последовательностей. Благодаря недавно завершенному проекту "Геном человека" известны аминокислотные последовательности всех белков в человеческом организме. Исследования по данному проекту также помогут в проектировании новых, не существующих белков. В случае успешного решения данных проблем мы сможем бороться с такими болезнями как рак, малярия, болезнь Альцгеймера, сибирская язва и другими генетическими и вирусными заболеваниями.[/quote]
http://www.boinc.ru/proect.htm#Rosetta
http://www.youtube.com/watch?v=GzATbET3g54 new!
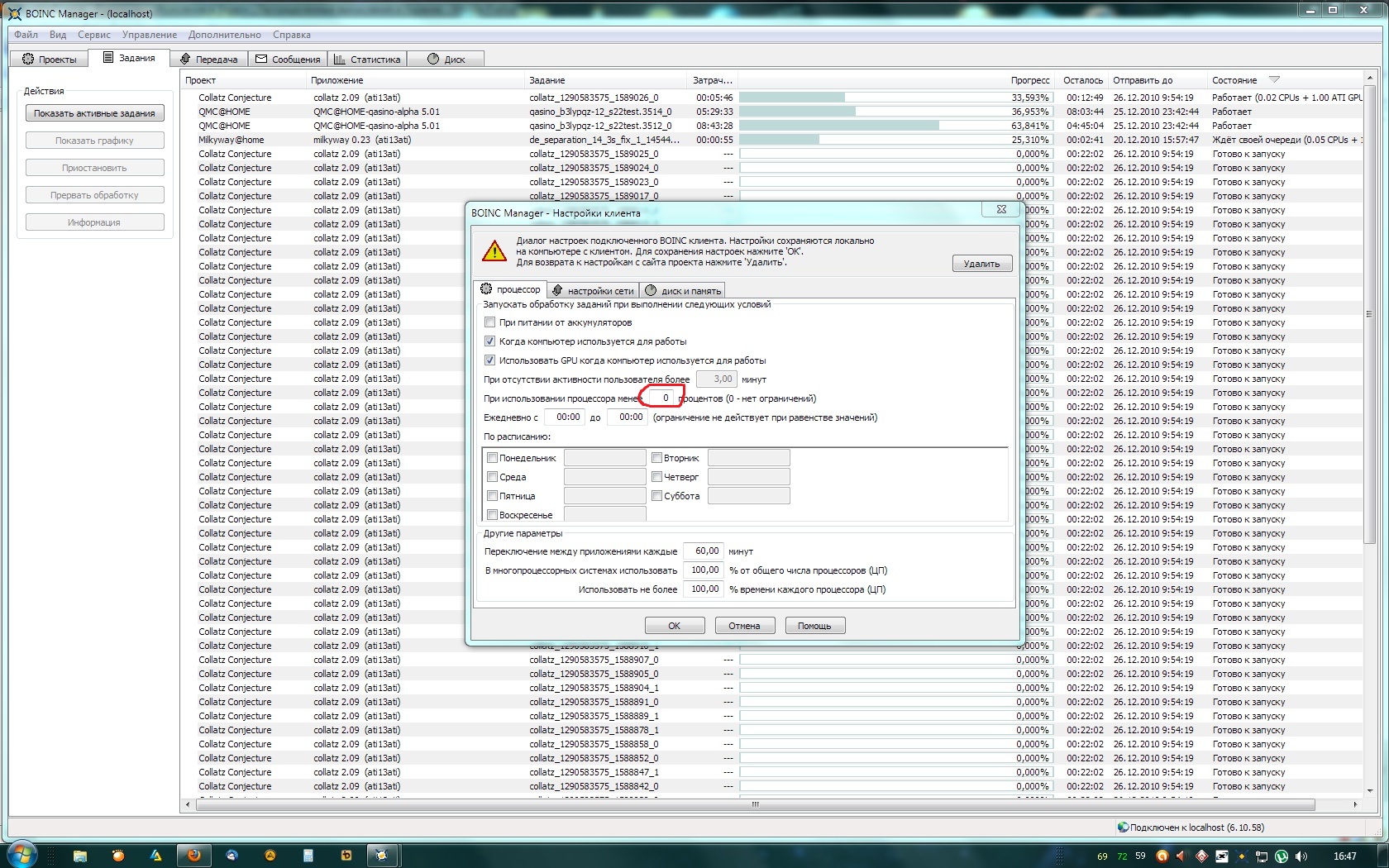
Для большей научной пользы выставьте время рассчета ВЮ - 1 день.
Для этого:
1. зайдите в Ваши настройки на сайте, http://boinc.bakerlab.org/rosetta/prefs.php?subset=project
2. выберите Target CPU run time = 1 day
3. нажмите Update
4. откройте БОИНК менеджер
5. если у вас сокращенный вид, нажмите Advanced View
6. выберите первую вкладку
7. выберите проект Розетта и нажмите кнопку "Обновить" сверху слева
8. Готово! Теперь каждая молекула будет "предсказываться" 1 день вместо 3 часов (по умолчанию)
http://www.boinc.ru/Doc/Roset/britt.htm
http://www.boinc.ru/Doc/Roset/FAQ/ScienceFAQ.htm
http://www.boinc.ru/Doc/Roset/FAQ/D_R_Research.htm
http://www.boinc.ru/Doc/Roset/atRosetta.htm
http://www.boinc.ru/Doc/Roset/FAQ/rosetta_FAQ.htm
http://www.boinc.ru/Doc/Roset/Active_WU.htm
http://www.boinc.ru/Doc/Bio_Diffs.htm
http://distributed.org.ua/forum/index.php?act=Attach&type=post&id=561
http://www.worldcommunitygrid.org/forums/wcg/viewthread?thread=10249&offset=0, боинк брать на оф сайте
График ППД команды за последние 60 дней:
Марка по теме проекта:

----------------------------------------------------------------------------------------------------------
Статус сервера выдачи заданий:

Автор: Arbalet May 29 2007, 12:31
Затея, на самом деле, очень хорошая и правильная!
Это настоящий бич боинк-проектов, что там тьма не очень сильных украинских команд. В результате ни о каких серьезных продвижениях в мировом рейтинге там можно и не мечтать. Другое дело, если бы большую часть удалось объединить в рамках проектов в одну команду... ![]()
И если кому-то удастся найти способ писать в личку участников боинк-проектов, тому можно купить ящик мороженого ![]()
Автор: Panda May 29 2007, 15:18
Хмм... Чи то я старий, чи "новое поколение выбирает...", але в наші часи кранчерів агітували на пиві... ![]()
Автор: Rilian May 29 2007, 21:37
Если что мы напишем админам статистики или регистрации письмо с просьбой выслать список мыл капитанов команд или учаснегов для дальнейшего их присоединения. Думаю ничего страшного в этом нет.
Скажите куда и кому написать письмо, я напишу (на англ)
Автор: nikelong Jun 1 2007, 06:09
Если что мы напишем админам статистики или регистрации письмо с просьбой выслать список мыл капитанов команд или учаснегов для дальнейшего их присоединения.
Я думаю тебе такого не дадут
Автор: Rilian Jun 1 2007, 15:17
как админу команды могут и прислать.
Автор: Rilian Jun 16 2007, 14:04
Подключился к проекту. Посчитаю несколько дней
Автор: uNiUs Jun 17 2007, 23:17
Да, распылятся, пожалуй, не стоит. Если есть желание представлять какую-то фирму или банк - незачем создавать отдельную команду, можно просто создать одну учетную запись и под ней считать всем из них, кто пожелает. Так у нас, например, сделал Profi-P(Profi-press) - это вроде как полиграфическая фирма. Только как связаться с капитанами остальных команд - ума не приложу... ![]()
Автор: Rilian Jun 17 2007, 23:27
так как ты капитан команды, довольно большой, я считаю что у тебя есть право связаться с админами проекта и попросить емейлы капитанов других украинских команд..
Автор: uNiUs Jun 18 2007, 00:08
так как ты капитан команды, довольно большой, я считаю что у тебя есть право связаться с админами проекта и попросить емейлы капитанов других украинских команд..
Думаю, не все так просто... И твое мнение и мнение админов проекта может не совпадать
Автор: Vladus Jun 18 2007, 01:20
На данный момент самые большие показатели в этом проекте у команды UKRAINE.
Может все вместе обсудим и решим в какую одну команду объедениться?
А участникам других команд выслать приглашение.
Что вы думаете по этому поводу?
Полностью поддерживаю эту идею!
Призываю всех желающих считать в Розетте присоединиться к команде UKRAINE.
Автор: nikelong Jun 18 2007, 06:31
А через БОИНК-профиля некоторых людей можно-ж выловить?
У меня так с парой человек получилось....даже в асе общался (!)
Автор: YuRi Jun 18 2007, 13:35
И через них выйти на капитанов их команд ![]()
Автор: uNiUs Jun 18 2007, 22:25
А через БОИНК-профиля некоторых людей можно-ж выловить?
У меня так с парой человек получилось....даже в асе общался (!)
Если есть хоть какие-то контакты в профиле типа мыла или аськи - то да, можно. Но к несчастью у большинства кранчеров нет даже самих профилей...
А еще было бы неплохо не только объединить украинские команды, но и попросить присоединиться тех, кто еще ни в одной команде не состоит. Таких тоже много - http://www.boincsynergy.com/stats/country.php?country=Ukraine&project=rah
Только вопрос, где взять контактную информацию, по-прежнему остается открытым...
Автор: ReMMeR Jun 18 2007, 23:05
погуглить по нику попытатся можно.
Автор: Vladus Jun 19 2007, 01:36
А еще было бы неплохо не только объединить украинские команды, но и попросить присоединиться тех, кто еще ни в одной команде не состоит. Таких тоже много
А также существуют.... непатриотичные
http://www.boincstats.com/stats/user_stats.php?pr=rosetta&st=0&or=&co=Ukraine&hosts=0
http://www.boincstats.com/stats/user_graph.php?pr=rosetta&id=100677
http://boinc.bakerlab.org/rosetta/forum_user_posts.php?userid=100677
Погуглить действительно иногда полезно:
http://forum.folding-community.org/profile.php?mode=viewprofile&u=10281
http://forum.autoua.net/showprofile.php?Cat=0&User=14217&Number=1283530&Board=carchoice&what=showthreaded&page=&fpart=&vc=1
http://forums.overclockers.ru/profile.php?mode=viewprofile&u=61939&sid=75b2373676852ad863fa05bcee9959f6
http://www.skoda-club.org.ua/index.php?option=com_forum&Itemid=26&page=viewtopic&t=3593
Похоже, что это все один человек....
И еще раз призываю помочь команде Ukraine, бо давно уже ближайшими перед нами в рейтинге находятся команды http://boinc.bakerlab.org/rosetta/top_teams.php?sort_by=total_credit&offset=100
Автор: Oleg82 Jun 19 2007, 05:13
а чувак и недогадывается что гдето на него папочку завели похлеще интерпола ![]()
Автор: uNiUs Jun 19 2007, 09:13
А также существуют.... непатриотичные
http://www.boincstats.com/stats/user_stats.php?pr=rosetta&st=0&or=&co=Ukraine&hosts=0
http://www.boincstats.com/stats/user_graph.php?pr=rosetta&id=100677
http://boinc.bakerlab.org/rosetta/forum_user_posts.php?userid=100677
Погуглить действительно иногда полезно:
http://forum.folding-community.org/profile.php?mode=viewprofile&u=10281
http://forum.autoua.net/showprofile.php?Cat=0&User=14217&Number=1283530&Board=carchoice&what=showthreaded&page=&fpart=&vc=1
http://forums.overclockers.ru/profile.php?mode=viewprofile&u=61939&sid=75b2373676852ad863fa05bcee9959f6
http://www.skoda-club.org.ua/index.php?option=com_forum&Itemid=26&page=viewtopic&t=3593
Похоже, что это все один человек....
И еще раз призываю помочь команде Ukraine, бо давно уже ближайшими перед нами в рейтинге находятся команды http://boinc.bakerlab.org/rosetta/top_teams.php?sort_by=total_credit&offset=100
Браво! Целое досье завел!
Жаль только, что он не хочет бросать TSC! Russia. Я с ним уже беседовал по этому поводу по аське еще где-то месяца три назад. А так помощь была бы очень даже кстати... А действительно за Украину обидно. (Особенно глядя на "Gay USA"
Автор: Vladus Jun 19 2007, 09:36
а чувак и недогадывается что гдето на нето папочку завели похлеще интерпола
Дык, вся информация открытая, в свободном инетном доступе.
Жаль только, что он не хочет бросать TSC! Russia. Я с ним уже беседовал по этому поводу по аське еще где-то месяца три назад. А так помощь была бы очень даже кстати...
Да, жаль.... Но есть еще несознательные/непроинформированные считающие. Хотя, подобное "досье" можно собрать только на активно пишущих в инете...
Автор: Rilian Jun 22 2007, 08:07
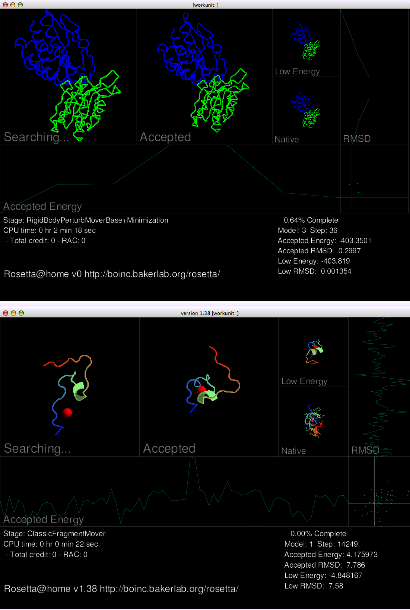
Что означают красные точки на графике?
http://i19.tinypic.com/6cs0ope.png
Минимальные состояния с предыдущих моделей ?
(За 24 часа предсказаний мой клиент успевает рассмотреть около 120 моделей)
Автор: uNiUs Jun 22 2007, 14:20
Что означают красные точки на графике?
http://i19.tinypic.com/6cs0ope.png
Минимальные состояния с предыдущих моделей ?
(За 24 часа предсказаний мой клиент успевает рассмотреть около 120 моделей)
Это цитата из http://boinc.bakerlab.org/rosetta/rah_graphics.php:
Последнее поле, в правом нижнем углу, составляет график энергии и RMSD каждого принятого перемещения. Они - тот же самый вид графиков, который показан на странице лучших предсказаний. Но в отличие от этого, Вы видите их для каждого *принятого* перемещения в течение траектории. Графики на http://boinc.bakerlab.org/rosetta/rah_top_predictions.php составлены только из сворачиваний с низкой энергией от каждой траектории.
Думаю, красными точками обозначены действительно минимальные состояния энергии. Хотя у меня таковых нет - только зеленые. Может, красными обозначены только наиболее удачные с точки зрения Розетты?
P.S: На моем древнем домашнем AthlonXP за 24 часа считается около 75 моделей. Хотя это еще зависит от количества шагов(step) обсчета, так что ориентироваться на эту величину как на показатель производительности не стоит.
Автор: Rilian Jun 22 2007, 14:35
у меня почти всегда когда я смотрю на графическое представление при подсчете Розетты, видны по несколько красных точек.
Допустим это минимальные значения. Надо только понять, это мин значения среди рассмотренных в данной ВЮ моделей, или среди всех ? Я спрашиваю, потому что иногда у меня зеленые точки "заходят" под красные, то есть находится меньшая энергия
Автор: uNiUs Jun 22 2007, 14:50
Сложно сказать. Попробуй посмотреть, не меняются ли расположение красных точек при переходе на следующую модель обсчета - и тогда все станет ясно. А вообще, теоретически, чем ниже и левее находится конкретная точка, тем лучше.
Автор: Rilian Jun 22 2007, 15:09
Вот пример, когда красные точки "выше" зеленых:
http://i16.tinypic.com/52c7jfr.png
Автор: Rilian Jun 27 2007, 18:01
Вышла новая версия клиента 5.70beta, которую мой БОИНК менеджер успешно скачал (2.85МБ) и установил
Автор: Rilian Jun 28 2007, 11:09
uNiUs, ты можешь объединить украинские команды?
http://boinc.bakerlab.org/rosetta/team_lookup.php?team_name=ukr&search=Search
при просмотре команд есть кнопка Initiate transfer
Автор: uNiUs Jun 29 2007, 09:33
uNiUs, ты можешь объединить украинские команды?
http://boinc.bakerlab.org/rosetta/team_lookup.php?team_name=ukr&search=Search
при просмотре команд есть кнопка Initiate transfer
У тебя во всех командах активна эта функция?
У меня везде активно только Join this team. А Initiate transfer - это смена капитана команды, а не объединение команд. Наскалько мне известно, команды нельзя объединять, даже если являешься капитаном обоих.
Автор: Rilian Jun 29 2007, 09:42
По ходу трансфер внутри команды возможен только если ты не капитан, а участник команды.
Но капитан может отсылать емейл всем участником. Поэтому надо сделать N ников, вступить во все маленькие команды и связаться с капитанами
Автор: uNiUs Jul 2 2007, 23:03
По ходу трансфер внутри команды возможен только если ты не капитан, а участник команды.
Но капитан может отсылать емейл всем участником. Поэтому надо сделать N ников, вступить во все маленькие команды и связаться с капитанами
Каким образом связаться с капитанами? Через трансфер? Очень оригинальный способ. Только написать таким образом ничего нельзя - можно только послать запрос на его мыло, которое так и останется неизвестным. Нет никаких гарантий, что трансфер молча будет отклонен без всяких вопросов.
Вообще не думаю, что над этим стоит ломать голову. Из всех перечиленных тобой команд хоть что-то представляет собой только BOINC@Ukraine, и то там сейчас активность близка к нулю.
Автор: uNiUs Aug 7 2007, 23:51
Это по поводу использования результатов Розетты проектом WCG (Хотя это итак известно ![]() )
)
Только не совсем понятно насчет версии. В Розетте сейчас 5.68-5.73. А WCG использует 5.18. 
Автор: KoDAk Aug 11 2007, 22:20
Похоже, что это все один человек....
ДА это я
только
"Профиль 2" не мое.
хмм ...
и обмолвились незлым тихим словом...
та я тож за обьединение...
а нас читанное как ? с нуля что ли ?...
.......
-----------
таки тут можно обьединятся без патери личной статистики )))))
тока что-то
от моего перехода пока непоменяласьстатистика команд
Автор: KoDAk Aug 12 2007, 08:07
"Попугаи набранные за одну команду не перезачитываются, а остаются в той команде на которую они были насчитаны "
НО САМОЕ ГЛАВНОЕ - СОХРАНЯЕНТСЯ ЛИЧНАЯ СТАТИСТИКА
И как итог объединить УКР команду технически возможно.
Автор: Tamagoch Aug 12 2007, 08:17
возможно объединить будущее укр. команды, но не прошлое.... разве плохая альтернатива?
Автор: KoDAk Aug 12 2007, 16:40
Та главное, что бы пошел процесс объединения (Так сказать консолидации)...
и кстати радуемся т.к. я уже ТУТ . ![]() .
.
из еше ценных кадров (ничейных)
есть тока
http://www.boincsynergy.com/stats/boinc-stats.php?id=10216&project=rah
и http://www.boincsynergy.com/stats/boinc-stats.php?id=192084&project=rah
все отальные на данный момент мертвые души...
Автор: Vladus Aug 13 2007, 09:38
Похоже, что это все один человек....
ДА это я
только
"Профиль 2" не мое.
хмм ...
и обмолвились незлым тихим словом...
ВАУ! С прибытием! И ссори за "досье".
Автор: KoDAk Aug 13 2007, 09:44
в правом нижнем углу твоего поста есть

Автор: Vladus Aug 13 2007, 09:58
в правом нижнем углу твоего поста есть

Сейчас есть..... А ведь небыло! И сейчас под инфой об юзере три кнопки, а недавно было две - небыло "Card". Хрень какая-то.... Я подозреваю, что у меня просто недогрузились картинки.....
p.s. ссори за оффтоп.
Автор: KoDAk Aug 13 2007, 19:23
вот уже на
95 месте
и 49 по скорости (ну это еше не вся моя скорость еше видна)
но всеравно жалковатые результаты (
Автор: KoDAk Aug 13 2007, 20:28
ВАУ! С прибытием! И ссори за "досье".
ТА даже любопытно )))
Автор: uNiUs Aug 14 2007, 03:38
Приветище! Не ожидал твоего появления в команде, хотя оч приятно! С вступлением в нашу скромную команду! ![]()
![]()
ЗЫ: А твой комп отжигает не по деццки! 300к! Редкий PC достигает такого рубежа, тем более в такой короткий срок. ![]()
ЗЗЫ: К сожалению, не знаю как связатся с max4dog или ndl. Первого асю вроде нашел, но он не отвечает.
Автор: KoDAk Aug 15 2007, 19:27
uNiUs,
можешь добавить мою аську в список на главной странице
http://distributed.org.ua/
Автор: nikelong Aug 16 2007, 15:52
KoDAk,
Такой вопрос не Униусу нужно задавать ![]()
Автор: KoDAk Aug 16 2007, 21:22
ну если знаешь кому так напиши , и я напишу тому кому надо.
Автор: ReMMeR Aug 16 2007, 21:32
KoDAk,
В Поддержку розетты ?
Автор: KoDAk Aug 17 2007, 21:40
ура с нами
max4dog
он сам или его кто то направил?
Автор: KoDAk Aug 17 2007, 22:16
-OIeksandr потенциальный активист (но пока не с нами) (
из слегка активных
-jeck (
- Sergey Stychynsky (
-
Автор: uNiUs Aug 18 2007, 23:54
Из тех украинцев, кто более-менее считает Розетту, но еще ни в какой команде не состоят :
jeck, Pavlo Narozhnyy, GNEEOT, OIeksandr, Igor Unzhakov, Woland, dad, Sergey Stychynsky, ndl, Anatoliy_SHUBA, Ayane, dukem.
Если кто-нить знает, как с ними связаться - отпишитесь, или сами намекните ![]()
А в общем - команда считает как никогда хорошо. Хотя надеюсь, что это еще не предел...
Можно даже сказать, что в Розетте Ukraine одна из самых сплоченных команд среди крупных проектов по боинку. ![]()
Автор: KoDAk Aug 19 2007, 00:36
вот мы и на 93 месте
Автор: Oleg82 Aug 19 2007, 05:30
вот мы и на 93 месте
также все достижения бей в традиционную тему ЙО!
Автор: x3mEn Aug 19 2007, 16:34
в мене Rosetta більше не роздає завдань... ![]()
![]()
Автор: uNiUs Aug 19 2007, 17:10
в мене Rosetta більше не роздає завдань...
Можливо, ти випадково натиснув кнопку "Не запрашивать новые задания". Краще скопіюй лог, наприклад в такому вигляді:
--------------------------------------------------------------------------------------------------------------------
19.08.2007 18:16:05|rosetta@home|Sending scheduler request: Requested by user
19.08.2007 18:16:05|rosetta@home|(not requesting new work or reporting completed tasks)
19.08.2007 18:16:10|rosetta@home|Scheduler RPC succeeded [server version 509]
19.08.2007 18:16:10|rosetta@home|Deferring communication for 4 min 2 sec
19.08.2007 18:16:10|rosetta@home|Reason: requested by project
--------------------------------------------------------------------------------------------------------------------
Автор: x3mEn Aug 19 2007, 18:06
в мене Rosetta більше не роздає завдань...
Можливо, ти випадково натиснув кнопку "Не запрашивать новые задания". Краще скопіюй лог, наприклад в такому вигляді:
та ні, цей варіант виключено, я вже перевіряв.
останній лог від rosetta такий:
19.08.2007 13:59:11|rosetta@home|Sending scheduler request: To report completed tasks
19.08.2007 13:59:11|rosetta@home|Reporting 1 tasks
19.08.2007 13:59:16|rosetta@home|Scheduler RPC succeeded [server version 509]
19.08.2007 13:59:16|rosetta@home|Deferring communication for 4 min 2 sec
19.08.2007 13:59:16|rosetta@home|Reason: requested by project
раніше, десь на 60% попереднього завдання, проект починав сам скачувати ще одне завдання. а тут, дивлюсь, не качає. окрім того faah скачує і обраховує свої завдання вправно.
вилікувалось Перезапуском проекта
хм... якщо таке буде повторюватись, від rosetta треба буде відмовитись,
як це було з Folding@Home - двічі обчислював завдання і двічі наприкінці підвисав комп, а після перевантаження результати обчислення злітали на 0. Може і збіг обставин, але я вирішив більше долю не випробувати.
тепер ось faah з місяць працює - політ нормальний.
Автор: uNiUs Aug 19 2007, 18:07
та ні, цей варіант виключено, я вже перевіряв.
останній лог від rosetta такий:
раніше, десь на 60% попереднього завдання, проект починав сам скачувати ще одне завдання. а тут, дивлюсь, не качає. окрім того faah скачує і обраховує свої завдання вправно.
Спробуй http://hhttp://boinc.bakerlab.org/rosetta/prefs.php?subset=global збільшити кеш завдань (Connect to network about every
(determines size of work cache; maximum 10 days)) до 2-х днів і натисни кнопку "Обновить".
Автор: x3mEn Aug 19 2007, 19:26
та ні, цей варіант виключено, я вже перевіряв.
останній лог від rosetta такий:
раніше, десь на 60% попереднього завдання, проект починав сам скачувати ще одне завдання. а тут, дивлюсь, не качає. окрім того faah скачує і обраховує свої завдання вправно.
Спробуй http://hhttp://boinc.bakerlab.org/rosetta/prefs.php?subset=global збільшити кеш завдань (Connect to network about every
(determines size of work cache; maximum 10 days)) до 2-х днів і натисни кнопку "Обновить".
в мене стоїть 0.3
останні 2 тижні я налаштувань не міняв, тому і дивно, що таке сталось.
Автор: uNiUs Aug 20 2007, 04:05
в мене стоїть 0.3
останні 2 тижні я налаштувань не міняв, тому і дивно, що таке сталось.
Якщо стоїть кеш 0.3 - тоді зовсім не дивно.
Автор: KoDAk Aug 20 2007, 19:35
У меня аналогично вычисления 24 часа кеш на 3 дня
Автор: x3mEn Aug 20 2007, 23:27
Якщо стоїть кеш 0.3 - тоді зовсім не дивно.
Це все дуже добре, але до цього ж працювало майже 2 тижні. І зараз працює, і завантажує, а тоді не завантажувало навіть за примусового Оновити та інші шаманства. Тому це не аргумент.
Інтернет в мене постійний, проблем немає. Окрім того інший проект свої завдання того самого часу завантажував.
Врешті, якийсь ґлюк. Буду сподіватись, що не системний.

Автор: uNiUs Aug 21 2007, 01:05
Виходячи з твого логу я не бачу навіть спроб Розетти зв'язатися з сервером(один із варіантів - кнопка Оновити). Твій кліент тільки відзвітував про обраховану ВЮ. Ніяких запросів на нові ВЮ, навіть не було синхронізаціі. Якщо хочеш розібратись - виклади весь лог мені в ICQ. Доречі добра новина - твоє завдання успішно зарахувалося, нові закачуються, тому не бачу причин для хвилювання. В мене кілька машин з досить поганим каналом інтернету працюють в Розетті з вищеописаними налаштуваннями працюють з листопаду 2006р - вищезазначених проблем не було. Доречі, на головній сторінці нашої команди я додав монітор статусу проекту. Можливо, це й зайве, бо сервери Розетти завжди працюють як годинник за виключенням досить коротких проміжків часу, відведених для профілактики серверів. Але таке буває нечасто, триває лише кілька годин та про це завжди можна прочитати на головній сторінці проекту.
Питання:Невже так важко виставити більший кеш? Хоча б до 1? Можеш мені повірити - для стабільності проекту це буде краще. ![]()
Автор: x3mEn Aug 21 2007, 19:52
Питання:Невже так важко виставити більший кеш? Хоча б до 1? Можеш мені повірити - для стабільності проекту це буде краще.
А навіщо? Я приймаю участь у двох проектах, інтернет в мене є постійно. Навіть якщо припустити, що деякий час інтернету не буде, комп без роботи не залишиться, тому що буде обраховуватись завдання іншого проекту. Оскільки середній час обчислення одного завдання 3-5 годин, то це означає, що інтернету має не бути десь близько стільки ж. А в мене такого практично не було. Більш того, на тижні комп в мене працює в середньому тих самих 5год. на день, тому навіщо мені пачка завдань на 24 год. обрахунку? :-/
Зараз все ґаразд, як тільки минає 50-60 %% минулого завдання, шедулер скачує наступне завдання. Що стосується того випадку, то проект завдань не скачував близько 24 години. Як тільки я дав команду Перезапустить проект, відбулась заміна модулів і одразу було скачано завдання. І до досі все нормально. Тому ніякого зв'язку з поняттям кешу я не бачу.
Автор: KoDAk Aug 27 2007, 19:17
с версии 5.10.20 менеджер стартует тихо в трей
Автор: KoDAk Sep 2 2007, 16:15
а с
"C:\Program Files\BOINC\boincmgr.exe" /s
всегда в трее
даже при ручном запуске
Автор: Rilian Sep 18 2007, 11:49
We're experiencing another hardware problem that is effecting the overall project, causing the validator and assimilators to fall behind. The temporary fileserver to which we've switched has dropped on of its RAID disks. We've found a replacement and the RAID is rebuilding now. Once finished the project should start catching up w/ itself...
Мы столкнулись с другой аппаратные проблемы, осуществление общего проекта, что вызывает одобрение и assimilators для отстающих. Временной файл-сервер, к которому мы перешли упал на ее RAID дисков. Мы обнаружили, замену и RAID восстанавливает сейчас. После завершения проекта следует начинать догнать сама ...
Автор: ReMMeR Sep 18 2007, 20:26
О Великий мастер ЙЙоДа !
Автор: Rilian Oct 2 2007, 00:26
Добавил PROMO VIDEO в шапку )
Автор: KoDAk Oct 9 2007, 20:52
3 000 000 на счету
нашей команды. УРА товарищи ![]() !!!!!!!
!!!!!!!
Автор: nikelong Oct 9 2007, 22:10
Символично!
ФАХ сегодня 30 000 000 набрал
Росета - 3 000 000 ![]()
Автор: Rilian Oct 9 2007, 22:32
И форум сегодня набрал 19000 сообщений! ![]()
![]()
Автор: uNiUs Oct 9 2007, 23:29
И в розеттовской команде Ukraine 100 учаснегофф... ![]()
Автор: KoDAk Oct 13 2007, 21:20
мой комп добрался до ТОП 80
http://boinc.bakerlab.org/rosetta/top_hosts.php?sort_by=total_credit&offset=60
Автор: KoDAk Oct 18 2007, 23:26
Change Log:
http://boinc.berkeley.edu/dev/forum_thread.php?id=1668
Oct 17, 2007
An article about Rosetta@home is in Nature. Congrats and thank you to all the volunteers that made this possible!
http://www.nature.com/news/2007/071016/full/449765a.html
Автор: Rilian Oct 18 2007, 23:58
Классная статья. Спасибо
Автор: KoDAk Jan 1 2008, 04:02
Опрос участников BOINC-проектов
http://boinc.berkeley.edu/poll.php
Автор: langolierra Jan 8 2008, 15:25
67 место?
Автор: KoDAk Jan 8 2008, 20:39
да уже дано, в прошлом году ))))
Автор: Rilian Jan 14 2008, 20:45
Перевел все 3 статьи на украинский язык и составил все в виде реферата
Отправил Реммеру, он выложит на сайте
Автор: KoDAk Jan 18 2008, 09:00
http://boinc.bakerlab.org/rosetta/top_teams.php?sort_by=total_credit&offset=60
Автор: KoDAk Feb 9 2008, 22:57
![]() http://boinc.bakerlab.org/rosetta/top_teams.php?sort_by=total_credit&offset=60
http://boinc.bakerlab.org/rosetta/top_teams.php?sort_by=total_credit&offset=60 ![]()
Автор: KoDAk Apr 12 2008, 20:49
http://www.apple.com/rosetta/
Автор: Alexei_Komarov Apr 17 2008, 19:58
http://www.boincsynergy.com/stats/country.php?sort=RC&project=rah
Автор: Rilian Apr 19 2008, 13:47
Реферат по розетте на украинском языке, если кому-то нужно ![]()
Приєднані файл(и) rosetta_referat.zip ( 630.87Кб )
Кількість викачувань: 1978
rosetta_referat.zip ( 630.87Кб )
Кількість викачувань: 1978
Автор: (_KoDAk_) May 14 2008, 18:25
тут http://boinc.bakerlab.org/rosetta/team_display.php?teamid=328
появиласся
Team message board for Ukraine
http://boinc.bakerlab.org/rosetta/forum_forum.php?id=67
Автор: Rilian Jul 6 2008, 20:55
А-а-а-а-а я не в топ 20 ![]()
Щя пофиксим
кстати, в шапке добавлено
http://www.worldcommunitygrid.org/forums/wcg/viewthread?thread=10249&offset=0, боинк брать на оф сайте
Автор: (_KoDAk_) Jul 7 2008, 22:25

Автор: ReMMeR Jul 8 2008, 09:41
Круто ! =)
Поздравления =) ![]()
![]()
Автор: fatum Jul 9 2008, 13:27
присоединяюсь к проэкту надюсь смогу помочь
Автор: Rilian Jul 9 2008, 13:46
присоединяюсь к проэкту надюсь смогу помочь
Вэлкам! (http://boinc.bakerlab.org/rosetta/show_user.php?userid=268004)
Советую выставить время работы одной ВЮ на "1 day" - для максимальной пользы
Для этого:
1. зайдите в Ваши настройки на сайте, http://boinc.bakerlab.org/rosetta/prefs.php?subset=project
2. выберите Target CPU run time = 1 day
3. нажмите Update
4. откройте БОИНК менеджер
5. если у вас сокращенный вид, нажмите Advanced View
6. выберите первую вкладку
7. выберите проект Розетта и нажмите кнопку "Обновить" сверху слева
8. Готово! Теперь каждая молекула будет "предсказываться" 1 день вместо 3 часов (по умолчанию)
Автор: (_KoDAk_) Jul 9 2008, 14:54
милости просим, будем вам рады.
Автор: (_KoDAk_) Aug 16 2008, 22:38
minirosetta application обновилась до 1.32
тепер в ней такая же графика что и была раньше в rosetta 5.хх
Автор: badman Sep 2 2008, 08:42
Хай пипл! Присоединяюсь к проекту. На первое место по RAC не замахиваюсь (пока ![]() ), а вот второе будет моим
), а вот второе будет моим ![]() . Кодак, подвинься
. Кодак, подвинься ![]()
Автор: nikelong Sep 2 2008, 08:52
badman,
Хорошие новости!
welcome aboard!
Автор: Rilian Sep 2 2008, 11:07
* abroad
Автор: mad Sep 2 2008, 13:20
* abroad
Ну тут кому куда больше нравится
Автор: Rilian Sep 2 2008, 14:49
Смените кто-нибудь линк тут
http://boinc.bakerlab.org/rosetta/team_display.php?teamid=328&sort_by=total_credit&offset=0
на http://distributed.org.ua/forum/index.php?showforum=26
Автор: Rilian Sep 8 2008, 13:07
У нас в проекте появилась http://boinc.bakerlab.org/rosetta/show_user.php?userid=276382 ![]()
![]()
Автор: Rilian Sep 17 2008, 01:53
Распределенными усилиями мозгов (_Kodak_) и rilian пофиксили описание команды на нашей оф страничке http://boinc.bakerlab.org/rosetta/team_display.php?teamid=328
Автор: (_KoDAk_) Oct 29 2008, 00:52
1 новость
The minirosetta application has been updated to version 1.39. In this version, we added two important applications to minirosetta, docking and protein folding with explicit zinc metal ion. New features have been added to the graphic to show chain colors for a multi-chain protein complex and display metal ions by sphere
2 єто баг на сайте интересная дата новой новости
Приєднані зображення
Автор: Rilian Oct 29 2008, 00:57
Кодак, лучшие предсказатели рассчитываются каждый день и висят в отдельном блоке выше основных новостей
Автор: (_KoDAk_) Oct 29 2008, 01:09
ну а почему тогда сегодняшняя новость датирована 10 иЮня ?
предыдущая новость была Oct 18, 2008
Автор: Rilian Oct 29 2008, 01:15
Наверное при создании новости они вводят дату вручную, и получилось не 10/6 а 6/10
Автор: (_KoDAk_) Oct 29 2008, 01:18
но сегодня 29.10.2008 !""!!!""!
Автор: Death Oct 29 2008, 14:49
то есть 10/29/2008?
Автор: (_KoDAk_) Oct 30 2008, 08:30
вот они и исправили дату
Автор: (_KoDAk_) Oct 31 2008, 21:47
появилось новое расчетное ядро Rosetta Mini with new score terms 1.02
про него David Baker сказал:
I'm also excited because I've been working on improvements to the energy function which underlies most rosetta@home calculations, and the first tests of the improvements should run on rosetta@home this next week!
We have many exciting calculations just starting to run on rosetta@home! If you split your computational resources between several different projects this would be a good time to temporarily increase rosetta@home's share. We are testing many new methods (see Chu's post today about zinc binding proteins for one of them) and we are eagerly examining each new work unit coming back from your computers. I am very excited personally as I'm doing the first large scale test of (what I think are) improvements in the energy function underlying the searches that I've been working on the past few months (these are the work units with "split" in them).
Мы только что получили хорошие новости из CASP организаторов - наши результаты были одними из лучших на casp8 и мы попросили описать метод мы использовали - в том числе все ваши работы - на CASP8 заседании в декабре. Есть ряд оценок исполнении автоматическому серверов на CASP8 доступны в Интернете, и сравнение наших моделей для серверов, показывает, что все Ваши усилия привели в среднем немного лучше моделей. Конечно, мы пока не знаем, других "человеческих" группы сделал CASP8 задач - для этого нам придется ждать встречи!
Я также рады, потому что я уже работал на улучшение энергетической функции, которая лежит в основе большинства Rosetta @ Home расчеты, и первые испытания улучшения должны работать на Rosetta @ Home этом на следующей неделе!
У нас много интересных расчеты только начинают работать на Rosetta @ Home! Если разделить ваши вычислительные ресурсы между несколькими различными проектами, это было бы хорошим временем для временно увеличить Rosetta @ Home долю. Мы проводим тестирование множества новых методов (см. Чу должность сегодня около цинк обязательного белков для одного из них) и мы с нетерпением изучении каждой новой работой подразделения возвращаются из вашего компьютера. Я лично очень рады, как я делаю первые крупномасштабные испытания (то, что я думаю, являются) повышение энергетической функции, лежащие в основе поисков, что я работал на протяжении последних нескольких месяцев (эти работы подразделения с "сплит "В них).
Автор: Death Nov 11 2008, 22:13
реальные результаты. тваюмать как долго я этого ждал
Posted 7 Nov 2008 7:27:53 UTC
Please see Sarel's post today on protein-protein interface design about to start up on rosetta@home. Sarel is designing proteins to block the action of pathogens like cholera, and we are excited that with your help we can make inhibitors for many proteins causing disease. We are also starting large scale tests of brand new structure prediction methods, including some that I have been developing myself. This is a very exciting time at rosetta@home!
Автор: (_KoDAk_) Nov 15 2008, 21:03
November 14, 2008
Outage Notice: the project will be down for maintenance starting at 2:00 PST today.
On an unrelated note: WE ARE PLANNING TO INCREASE THE DEFAULT RUN TIME TO 6 HOURS AND THE MINIMUM TO 3 TO REDUCE THE LOAD ON OUR SERVERS. When and how this will occur has not yet been decided. If you have any concerns or would like to add to the discussion please post to this thread.
November 14, 2008
Our fileserver crashed last night. We are planning to upgrade the system very soon. Sorry for any inconvenience.
Автор: Zedus Nov 25 2008, 22:16
интересный прожект... И сюда внесу свою лепту ![]()
Плохо только, что задания такие "тяжелые" (
когда интернет по траффику, с этим приходится считаться...
Автор: (_KoDAk_) Nov 26 2008, 01:16
интересный прожект... И сюда внесу свою лепту
Плохо только, что задания такие "тяжелые" (
когда интернет по траффику, с этим приходится считаться...
высвляешь растектное время задания 24 часа и трафик становится меньше))
Автор: (_KoDAk_) Dec 27 2008, 00:34
такое событие пропустили
Jul 14, 2008
Predictor of the day: Congratulations to Admin (Team Ukraine) for predicting the lowest energy structure for workunit FR_298_relax_3292_0 !
Автор: tribal Jan 7 2009, 20:16
прикольненько - считается розета 6 с копейками часов, остается 20 минут и 3% соответсвено, потом бах - посчитано 4 с копейками часа и всего лишь 73%... и заново считаем ![]() ))
))
Автор: Rilian Jan 7 2009, 20:17
какая версия проги? чето пишет БОИНК, что пишет розетта?
Автор: tribal Jan 7 2009, 23:17
боинк 6.4.2.... а никто и не ругалси.. просто он его остановил и перезапустил почему-то с меньших процентов... вопщем боинк у мя живет своей жизнью
Автор: Rilian Jan 7 2009, 23:45
ненене должен быть подробный лог в БОИНКЕ. скопируй все что есть
Автор: tribal Jan 8 2009, 11:50
Rilian,
да фиг с ним, вчера просмотрел весь лог ниче кроме paused и restart про розету не было... досчитался и хорошо, главно что очки начислили ![]()
Автор: Rilian Jan 8 2009, 12:47
tribal, ну ок
пока вцг в отключке, считаю Rosett-у
Автор: tribal Jan 8 2009, 17:44
Rilian,
Автор: Rilian Jan 8 2009, 21:08
если ты подключен к проекту Human Proteonome Folding то да, там на алгоритма розетты фолдятся белки
Автор: (_KoDAk_) Jan 8 2009, 22:52
скачет процентов бывает если ты менял время расчета заданий
но они обычно пролоджает считать....
Автор: (_KoDAk_) Jan 11 2009, 15:39
так перебои с заданиями закончились
и вот что бывает если ошибится в одну букву
http://www.youtube.com/watch?v=W3aN6-xGjMw
Автор: (_KoDAk_) Jan 20 2009, 22:07

итак вынашиваются планы по выполнению расчетов на Xbox 360.
http://boinc.bakerlab.org/rosetta/forum_thread.php?id=1262&nowrap=true#29151
http://boinc.bakerlab.org/rosetta/forum_thread.php?id=4637
Автор: (_KoDAk_) Jan 30 2009, 05:14

![]()
![]()
Автор: nikelong Feb 14 2009, 19:24
Да сам видел Results ready to send 5,608
Но как бы эта цифра еще с прошлого года не висела ... ?
http://www.boinc-af.org/content/view/816/303/
http://www.pnas.org/content/104/37/14664.full.pdf
http://www.boinc-af.org/content/view/767/303/
http://www.boinc-af.org/content/view/247/282/
http://www.boinc-af.org/content/view/26/219/
http://www.dp.by/wiki/Projects/Scilinc
http://www.dp.by/wiki/Projects/Rosettaathome
http://www.irelandboinc.com/projects/40-projects/110-rosettahome
http://boinc.bakerlab.org/rosetta/rah_about.php
Автор: (_KoDAk_) Feb 15 2009, 20:28
February 16, 2009
A fileserver/SAN hiccup caused the entire project to pause for a number of hours. Sorry for the outage. -KEL
Автор: Rilian Mar 3 2009, 15:20
Немного перефразирую свой 2 вопрос.
В проэкте Rosetta мне за одно выполненое задание начисляют от 20-35 очков. Посмотрев статистику других участников я увидел что им начисляется до 60 очков. От чего это зависит?
Нужно выставить длину задания в максимум, например 1 день.
Target CPU run time
http://boinc.bakerlab.org/rosetta/prefs.php?subset=project
Автор: Rilian Mar 12 2009, 23:33
Недавно Дэвид Бэйкер сообщил что его команда пишет аж 7 статей о том что они проделали за последнее время
Вообще, можно оч много интересного почитать в его журнале
![]() http://boinc.bakerlab.org/rosetta/forum_thread.php?id=1177
http://boinc.bakerlab.org/rosetta/forum_thread.php?id=1177
![]()
Автор: (_KoDAk_) Sep 11 2009, 10:46
September 10, 2009
The validator and scheduler servers are currently slowly processing a large work unit. We have reprioritized the WU after finding that it is causing server problem. However, it will take a while for the existing jobs to clean out. Meanwhile, server lags are expected.
кароче ожидаются тормоза с сервером
Автор: Arbalet Jan 28 2010, 16:24
В проекте Rosetta@home добрались до больших белков и свиного гриппа
Проект распределенных вычислений Rosetta@home продолжает оправдывать вложенные в него вычислительные мощности добровольцев. От руководителя проекта Дэвида Бэйкера стало известно о публикации научной статьи в Science magazine, посвященной вычислительным методам, которые позволяют определять трехмерные структуры белков размером до 200 аминокислот. Кроме того, научный сотрудник лаборатории Бэйкера сообщил об успехах в поиске ингибиторов вирусного белка гемагглютинина, ответственного за заражение клеток организма вирусом гриппа.
Кому интересны подробности - http://prostonauka.com/v-Rosetta-home-dobralis-do-svinogo-grippa! ![]()
Автор: Death Feb 1 2010, 20:59
http://boinc.bakerlab.org/rosetta/forum_thread.php?id=4477&nowrap=true#65045
Hi all,
my name is Tim Whitehead and I've been a postdoc in the Baker lab for about a year. My projects deal with the prediction and design of protein-protein interfaces. In this thread, Sarel has explained the design problem for protein-protein interfaces. Progress in this area can drive the next generation of treatments against pathogens like Mycobacterium tuberculosis or Influenza.
I have been working with Sarel on the design and experimental characterization of designs that target disparate proteins on both pathogens. We have a couple of successes thus far, which we are extremely excited about, but need to have better and better designs to show the validity of our design approach.
In this thread, Sarel has explained why and how we are targeting Influenza by designing a protein inhibitor of a protein that is displayed on the surface of the virus.
For Tuberculosis, our task is slightly more difficult. TB is caused by a bacterium called Mycobacterium tuberculosis. The bacteria is encased in a waxy coating of mycolic acid, which hurts our body's ability to expell the bacterium (for more information, see http://en.wikipedia.org/wiki/Mycobacterium_tuberculosis).
Instead of designing an enzyme that could degrade the coating, we are targeting a protein that is involved in the `synthesis` of mycolic acid. You may have already seen BOINC descriptions that say "docking of designs against 2CGQ" - 2CGQ is the protein that we are trying to inhibit. I am hoping that your contribution from ROSETTA@HOME will give us the increased quality of our designs that we need. Thanks for the help!
strauch
Message 65126 - Posted 27 Jan 2010 6:34:38 UTC
My name is Eva-Maria Strauch and I am one of the post-docs in the Baker lab working on the design of protein-protein interactions. I am very excited about having the possibility to run on R@H, thanks to your generous contributions! Protein design requires a lot of sampling since there are so many many different possibilities to be considered carefully. This will work out now faster with your help!
I am currently designing proteins to bind to a model system which we use to calibrate our computational protocols. This particular target is the back end of a human immune-system antibody (Fc fragment, which is what you will see under the work unit descriptions http://en.wikipedia.org/wiki/Fc_region). The cool thing about Fc is that nature provides us with several proteins that bind to it, hence it taught us very important lessons on how to design possible binding proteins to it. We now want to see whether we've figured this one out and can mimic nature's ability to come up with binders towards this target. Success will further contribute to the optimization of our protocols to generate several protein-based inhibitors to attack cancer and infectious diseases. Questions we want to answere with this design project are: How well do we perform in comparison with nature? and if we understood all the information we got out of those native interactions, are we able to produce several de novo binding proteins that mimic the atomic interactions seen with native interaction Fc has with different proteins? This project will contribute to our understanding of how proteins recognize one another. Promising designs will be experimentally verified and also further characterized to see how well we performed, and what was possibly missing that we should include into our computational design protocol.
Thanks again for your contribution!
Автор: Rilian Feb 1 2010, 21:14
ооо, надо вернуться в этот проект, пока в WCG IADS-1 находится в стадии обработки результатов
Автор: corsar83 Feb 4 2010, 11:45
Перестал BOINC вчера запрашивать задания для проца, только для видеокарты, которых нет. У всех так или это у меня чото.
Автор: Gelo Apr 13 2010, 07:39
Новости
Apr 12, 2010
The minirosetta application has been updated to version 2.10. For details and to report bugs, go to http://boinc.bakerlab.org/rosetta/forum_thread.php?id=5294
Автор: re_SET Apr 13 2010, 20:26
Ребята! а ГПУ-клиент имеется на розетке?
Автор: Rilian Apr 13 2010, 20:34
Ребята! а ГПУ-клиент имеется на розетке?
http://boinc.bakerlab.org/rosetta/apps.php
Автор: Algon May 15 2010, 13:56
15.05.2010 14:54:10 rosetta@home Sending scheduler request: To fetch work. Requesting 84116 seconds of work, reporting 1 completed tasks
15.05.2010 14:54:15 rosetta@home Scheduler request completed: got 0 new tasks
Задания на сервере есть, кеш на сутки (задание считается меньше 3 часов).
Почему может быть?
Автор: corsar83 May 15 2010, 15:07
Вроде нормально все, последний раз скачал задание сёдня в 12. Может поставь в настройках розетта счёт на максимальные 24 часа. А то наверно 3 стоит.
Автор: corsar83 May 18 2010, 19:02
Чото глючная какаето версия розетта 2.14. Эти новые задания CASP, считает считает часами, потом в любой момент может остановится расчет, время идет, а прогрес не движется. Пошол я опять считать WCG.
Автор: gladiator_maximus May 19 2010, 15:42
про себя я этот проект назвал error@home.
слишком много заданий слетает в эррор.Пустая трата процессорного времени...
Автор: ShNurik May 19 2010, 15:53
розетта 2.14 отлично работает. скачивает.. отправляет.. ошибок не было ![]()
Автор: uNiUs May 19 2010, 23:29
про себя я этот проект назвал error@home.
слишком много заданий слетает в эррор.Пустая трата процессорного времени...
Специально проверил резалты своего разогнанного компа - из сотни заданий всего два в ошибках, один сабортился на 15-й секунде, другой на 16-й. О каких часах затраченного времени впустую речь? Дело, скорее, в конкретном компе (переразгон, перегрев, мало памяти, програмные глюки etc.)
Автор: gladiator_maximus May 20 2010, 06:24
uNiUs,
Проц не разогнан, ОЗУ 3 Гб, перегрева нету, в других проектах (космология,эйнстейн) задания в эррор не валились.
Вот посмотрите - http://boinc.bakerlab.org/rosetta/results.php?userid=378965 (ДОСТУП ЗАПРЕЩЕН)
из шести-два в эрроре, и это спустя несколько часов расчетов.
Автор: Rilian May 20 2010, 10:03
gladiator_maximus,
http://boinc.bakerlab.org/rosetta/results.php?hostid=1276956
в двух ошибочных заданиях на компе
[2010- 5-16 20:38:47:] :: BOINC:: Initializing ... ok.
Can't acquire lockfile - exiting
[2010- 5-16 20:39:28:] :: BOINC:: Initializing ... ok.
Can't acquire lockfile - exiting
значит проблемы с доступом на компе. антивирусами балуешься? диск чем-то занят?
Автор: gladiator_maximus May 20 2010, 15:43
Rilian,
антивир нод32 стоит,попробую в нем добавить розетту в список исключений.
хотя розетта не мой основной проект, её я периодически посчитываю.
Еще в этом проекте, ну не то чтобы напрягает, но не очень нравится то, что когда закончил считать вюху , розетта из памяти не выгружается, продолжая занимать ОЗУ, только ребут компа помогает освободить ОЗУ от розетты.
Автор: Rilian May 20 2010, 15:47
каким образом ты это вычислил?
вообще на оф форуме на каждую версию минирозетты есть топик, где нужно сообщать о багах
Автор: Algon May 20 2010, 16:55
А может в настройках Boinc просто стоит "Оставлять неактивные приложения в памяти"? Вот она и оставляет...
За время пентатлона (и после него) посчитал 104 задания, из них 2 вылетели с ошибками, остальные нормально посчитались.
Автор: gladiator_maximus May 20 2010, 17:05
Algon,
"Оставлять неактивные приложения в памяти" ,галочка на этом пункте не стоит и не была никогда активирована.
Rilian,
легко вычислил в Диспетчере задач.И что странно, каждое посчитанное задание остается в памяти,занимает мегабайт так 250.
В других проектах подобных странностей нет.
Автор: Rilian May 20 2010, 17:14
Algon,
"Оставлять неактивные приложения в памяти" ,галочка на этом пункте не стоит и не была никогда активирована.
Rilian,
легко вычислил в Диспетчере задач.И что странно, каждое посчитанное задание остается в памяти,занимает мегабайт так 250.
В других проектах подобных странностей нет.
покажи скриншот с боинка и одновременно с диспетчера задач.
не может быть что посчитанное задание где-то может висеть
Автор: gladiator_maximus May 20 2010, 21:48
Rilian,
как буду опять считать розетту, покажу скриншоты.
Автор: gladiator_maximus May 26 2010, 21:37
Вот скрин http://fastpic.ru/view/5/2010/0526/9e3efe9eaceb6e4562e43b79597360bb.jpg.html
Задания благополучно ушли в эррор, и так же благополучно висят в ОЗУ.
Минирозетта 2.14. добавлена в список исключения Нод32. Дело не в антивире значит.
Тогда в чем?
Автор: Rilian May 26 2010, 21:45
что-то явно помешало процессу
попробуй день не качать новый софт, отключи нод, и запусти розетту еще раз.
кстати, ты запускаешь боинк от другого юзера. проверь его права на запись в тех папках
Автор: gladiator_maximus May 27 2010, 06:30
Rilian,
В Диспетчере задач имя пользователя проектов от БОИНКА те же, что и папка в которой они находятся.
Как можно проверить права на запись?
А что значит "не качать новый софт"?
Автор: uNiUs May 28 2010, 07:42
gladiator_maximus,
можно поступить проще - просто запусти boincmgr.exe под админом и посмотри, повторится ли проблема.
А антивирь случайно не ESET NOD32 Smart Security?
ЗЫ: Если ось - ХР проблема может быть в лимите на 10 коннектов, особенно учитывая кучу запущенного софта + торренты.
Автор: xapaktep May 28 2010, 11:20
У мене теж нещодавно почалися якісь трабли. Раніше на завдання йшло до 6 годин - а тепер від 30 до 70 годин. Жодних змін в залізі не відбувалось. При цьому іноді час йде, а процент завдання не змінюється. Хелп!
Автор: corsar83 May 28 2010, 12:05
6 часов это потому, что в профиле стоко стоит. Максимум можно 24 часа поставить. Но у меня на версии 2.14 тоже такая же фигня. Время идет, а процент не увеличивается. Задание просто какбы перестаёт считатся. Помогает пауза и снова запуск. Но постоянно тыкать это слишком ![]()
Автор: gladiator_maximus May 28 2010, 16:24
uNiUs,
Антивир Нод32 не Smart Security, обычный , фаер Комодо 4.0, минирозетту тоже добавил в список разрешенных приложений.
Лимит у меня на 100 коннектов.
Вот уже 15 часов считается еще одно задание-посмотрим,отпишусь.
Автор: Rilian May 30 2010, 05:28
uNiUs,
Антивир Нод32 не Smart Security, обычный , фаер Комодо 4.0, минирозетту тоже добавил в список разрешенных приложений.
Лимит у меня на 100 коннектов.
Вот уже 15 часов считается еще одно задание-посмотрим,отпишусь.
минирозетту добавил только сейчас?
с виду, задание досчиталось успешно http://boinc.bakerlab.org/rosetta/results.php?hostid=1276956
Автор: gladiator_maximus May 30 2010, 08:43
Rilian,
В список разрешенных приложений Комодо минирозетту и базы добавил как раз перед просчетом этой успешной вюхи.
Может случайность, а может и нет.
Щас космологию считаю, завтра к вечеру досчитаю, перейду на розетту-посмотрим.
Автор: gladiator_maximus Jun 1 2010, 19:04
Еще два задания успешно засчитались.
Видимо все таки фаервол порол задания.
А может и нет. Посмотрим.
Автор: Rilian Jun 2 2010, 09:11
Еще два задания успешно засчитались.
Видимо все таки фаервол порол задания.
А может и нет. Посмотрим.
а все начиналось с того что "проект кривой"
Автор: gladiator_maximus Jun 2 2010, 15:34
Rilian,
Да ладно, с кем не бывает.
Главное найти корень проблемы и решить ее.
Автор: G.R.Max Jun 19 2010, 11:55
Странная ситуация.
Я Розету не считал уже год, тут решил опять её попробовать.
Раньше был в другой команде. Перевёл розету в вашу команду, и просчитал задание одно на сутки.
Странное дело, но на сайте http://stat.polarseti.ru/index.php?t=11&p=0 не появилась моя статистика.
Хотя Розета засчитала 183 поинта. По другим проэктам проблем не было, они высвечиваются, а розеты нету(((
Автор: Rilian Jun 19 2010, 19:11
G.R.Max, смотри статистику по BoincStats. Поларсети глючит, особенно в новых аккаунтах и при переходах из команд
Автор: corsar83 Aug 9 2010, 15:15
Aug 8, 2010
Rosetta@home related advances have been featured in the national media twice in the last month. The first advance was the design of a protein catalyst that joins two molecules in a way not found in Nature; this was reported in Science magazine in July. The second advance is the finding that FoldIt players can solve hard structure prediction problems; this was reported in Nature magazine this week. These advances were covered in the New York Times, the LA times, and likely in your local newspaper. Thanks to all of you for your contributions to Rosetta@home!
Автор: corsar83 Sep 22 2010, 10:05
Sep 21, 2010
The minirosetta application has been updated to version 2.15.
Проснулись после отпуска ![]()
upd: add new protocols for symmetrical oligomers and membrane proteins.
Автор: Brodyaga Oct 15 2010, 13:18
Как командное ППД за вчера вверх стрельнуло. WCG отвалился, так все, кто заданиями не запасся, сюда перешли ![]()
Автор: Rilian Oct 28 2010, 21:23
2010-10-28: The minirosetta application has been updated to 2.17.
![]()
![]()
Автор: corsar83 Oct 29 2010, 12:49
2010-10-28: The minirosetta application has been updated to 2.17.
Чото на сайте ничо про 2.17 не сказано. Откуда нарыл инфу?
Автор: Rilian Nov 7 2010, 14:54
я юзер дня! ![]()
Автор: gladiator_maximus Nov 7 2010, 15:00
Rilian, Поздравляю!
Автор: Неадекватный Тролль Nov 19 2010, 11:19
Всем привет.
В настройках учетной записи на Rosetta@home ( http://boinc.bakerlab.org/rosetta/home.php ) Cross-project ID: 5b543316a264c56d02faf3d8c20081d0, но если пройти по ссылке на BOINC all Project Stats ( http://www.allprojectstats.com/showuser.php?id=5b543316a264c56d02faf3d8c20081d0 ), то там показывает совсем другого пользователя...
Почему так происходит, и какой же на самом деле у меня CPID?..
P.S. Электронная почта у меня одна и та же указана везде..
P.P.S. Запускал на 4-к компьютерах, но только один проект Rosetta@home, так что из-за разных проектов вроде не должно такого быть..
P.P.P.S. Так, ладно, зарегистрировался на http://www.boincsynergy.com, прописал туда свой CPID с Rosetta@home, жду 8 часов, написали, что он должен обновиться ниже написали, что CPID "is INCORRECT"..![]()
Странно, но теперь на http://boincstats.com прописался мой CPID (который, по идее, неправильный), раньше (при регистрации) там было пустое поле..
Автор: Rilian Nov 19 2010, 14:31
Неадекватный Тролль, почитай как соединить ЦПИДы http://distributed.org.ua/forum/index.php?showtopic=2476
Автор: uNiUs Nov 19 2010, 14:32
Подожди сутки-двое, тебя еще не подтянули сайты статистики.
Автор: Неадекватный Тролль Nov 19 2010, 14:47
Rilian, эту тему я нагуглил прежде всего. Просто непонятно было от чего отталкиваться: какой правильный CPID, сколько их вообще у меня оказалось и какие..
uNiUs, ок. А пока этого не произошло ![]()
Автор: Rilian Nov 19 2010, 16:51
Почему так происходит, и какой же на самом деле у меня CPID?..
посмотри в аккаунте на Розетте
Автор: Неадекватный Тролль Nov 19 2010, 19:33
Воо, по-крайней мере, на boincstats.com стало нормально отображаться ![]() http://boincstats.com/stats/boinc_user_graph.php?pr=bo&id=5b543316a264c56d02faf3d8c20081d0
http://boincstats.com/stats/boinc_user_graph.php?pr=bo&id=5b543316a264c56d02faf3d8c20081d0
И даже на DC появился http://stats.free-dc.org/stats.php?page=userbycpid&cpid=5b543316a264c56d02faf3d8c20081d0
Значит все наладится, спасибо за помощь ![]()
P.S. попробовал GPU-проект запустить, видеокарта с 43 градусов до 61 поднялась.. но стабильно 60-61.. жалко мне ее стало - отменил ![]() ведь в играх она хоть иногда отдыхает, а здесь ее жарят по-полной
ведь в играх она хоть иногда отдыхает, а здесь ее жарят по-полной ![]()
Автор: KING100N Nov 19 2010, 20:59
P.S. попробовал GPU-проект запустить, видеокарта с 43 градусов до 61 поднялась.. но стабильно 60-61.. жалко мне ее стало - отменил ![]() ведь в играх она хоть иногда отдыхает, а здесь ее жарят по-полной
ведь в играх она хоть иногда отдыхает, а здесь ее жарят по-полной ![]()
Не, 60 градусов - это еще не жарят. Вот >90 и 24/7 в таком режиме, вот это да, это другое дело. Правильно, пусть лучше и дальше отдыхает. Лучше оставить сегодняшний топ пылиться до лучших времен, а через пару-тройку лет продать за 400 грн.
Автор: Неадекватный Тролль Nov 19 2010, 21:32
Не, 60 градусов - это еще не жарят. Вот >90 и 24/7 в таком режиме, вот это да, это другое дело. Правильно, пусть лучше и дальше отдыхает. Лучше оставить сегодняшний топ пылиться до лучших времен, а через пару-тройку лет продать за 400 грн.
Не, не топ, всего лишь MSI N250GTS Twin Frozr Green Edition ( http://www.msi.com/index.php?func=proddesc&maincat_no=130&cat2_no=663&prod_no=1860 ).. Это же даже не ОС.. Я хоть и б/у брал, но видно, что вещь для себя бралась (такие штуки обожаю
Интересно, есть ли статистика об используемых таких "домашних" картах? Хотя бы в нашей команде?
Сори за оффтоп, такое ^^ можно здесь писать? Вроде тема не об этом, с другой стороны я только этот проект качаю
Автор: Rilian Nov 19 2010, 23:00
Неадекватный Тролль, нічего, оффтопік потом перенесем ![]()
http://distributed.org.ua/forum/index.php?showtopic=2133
Автор: Неадекватный Тролль Dec 9 2010, 06:55
сервер даун?
Автор: UA_Lex Dec 12 2010, 13:47
Сегодня решил попробовать себя в Rosetta (основной проект FAH). Подскажите пожалуйста, почему мне пришлось снизить загрузку проца до 80% (Athlon BE 7850 2.8Gh) чтобы он не переходил в Suspending computation - CPU usage is too high. Это получается, что проц начинает нестабильно считать? И еще, как отключить скринсейвер, нигде в настройках не нашел. Извините, если не в ту ветку написал. Заранее спасибо!
Вот щас снова клиент в suspend (на несколько секунд) ушел даже при 80% загрузке (
Автор: Gelo Dec 12 2010, 16:52
Сделай так
и можешь делать 100% загрузку ЦП
Автор: UA_Lex Dec 12 2010, 17:40
Спасибо, сделал. Почему то первое задание завершилось - Ошибка вычислений. В чем может быть причина?
Автор: Gelo Dec 12 2010, 18:22
Ошибки вычислений - это нормальное явление, если они одиночны. Вот когда они множественные тогда и надо искать причину( глючная память, хреновое питалово проца и тд.)
Автор: Rilian Dec 12 2010, 23:46
Спасибо, сделал. Почему то первое задание завершилось - Ошибка вычислений. В чем может быть причина?
причина написана в логе задания. посмотри на сайте и напиши сюда, будем разбираться
Автор: corsar83 Dec 16 2010, 13:29
Dec 10, 2010
Our article on protein energy landscapes done using @BOINC on Rosetta@HOME is out! http://www.miketyka.com/2010/12/energy_landscapes/
Автор: Неадекватный Тролль Jan 7 2011, 17:28
У них там каникулы чтоль?
Автор: pvtt Jan 25 2011, 07:03
хотів би дізнатись чому на одному компютері очків дає трохи більше ніж просить бонік, а на другому наоборот?
приклад: пентіум 4 claimed credit granted credit
394461435 360725447 24 Jan 2011 8:10:52 UTC 25 Jan 2011 3:06:27 UTC Over Success Done 7,819.14 12.09 20.33
394348334 360619138 23 Jan 2011 21:22:18 UTC 24 Jan 2011 21:25:53 UTC Over Success Done 12,788.65 19.77 29.34
394333683 360604852 23 Jan 2011 20:04:20 UTC 24 Jan 2011 21:25:53 UTC Over Success Done 18,998.85 29.37 44.01
амд семпрон 145 з розблокованим другим ядром
394627008 360881628 24 Jan 2011 22:52:19 UTC 25 Jan 2011 3:56:05 UTC Over Success Done 6,851.63 39.89 20.33
394555718 360813284 24 Jan 2011 16:33:08 UTC 25 Jan 2011 1:40:38 UTC Over Success Done 10,066.36 58.60 49.53
394221161 360495882 23 Jan 2011 10:03:58 UTC 24 Jan 2011 22:52:19 UTC Over Success Done 20,808.19 121.14 70.07
![]()
чомусь без пробілів вийшло
Автор: uNiUs Jan 25 2011, 14:39
хотів би дізнатись чому на одному компютері очків дає трохи більше ніж просить бонік, а на другому наоборот?
В Rosetta@home на claimed credit впливає лише boinc CPU benchmarks, в той час як на реальний час обчислення завдання впливає не лише потужнiсть процессору, а загальна швидкодiя зв'язки CPU - шина - RAM. Тому на ПК з швидкою RAM granted credit > claimed credit, i навпаки.
Автор: pvtt Jan 25 2011, 15:36
Зрозумів, дякую.
Автор: corsar83 Oct 7 2011, 12:25
Oct 6, 2011
Journal post from David Baker
A recent issue of Nature describes an exciting result from Rosetta@home in collaboration with the NMR spectroscopy laboratory of Lewis Kay in Toronto. Like almost all machines, proteins in order to carry out their functions have to move (change their conformation somewhat) but it has been extremely difficult to determine what these conformational changes are. Lewis Kay's group has developed new methods for getting experimental information on the higher energy very shortlived conformations proteins visit while carrying out their functions. This data is not sufficient to determine the structure of these "excited state" conformations using conventional methods. However, as the paper shows, we can use these experimental data to guide Rosetta and Rosetta@home structure calculations, and produce models of these states. We went one step further than this in the paper by using Rosetta design calculations to stabilize the excited state, and subsequent experiments confirmed the validity of the model. This combination of experimental NMR data, Rosetta structure calculations, and Rosetta design should be very powerful in understanding how proteins carry out their functions.
Автор: Rilian Oct 23 2011, 13:26
Rosetta@Home: Project News Oct 22, 2011
2011-10-22:http://boinc.bakerlab.org/rosetta/forum_thread.php?id=1177&nowrap=true#71460 from David Baker
Today's issue of Science magazine describes an exciting new approach to HIV vaccine design using Rosetta. In contrast with other viruses such as polio and influenza, inactivated HIV or HIV proteins have not worked as vaccines, and hence as you know there is currently no effective HIV vaccine. Our approach to vaccine design is to take the bits of the HIV surface protein that people make antibodies to, and using Rosetta graft them onto small stable scaffolds that can be made in large quantities and potentially could be useful as vaccines. We've shown earlier that this can be done straightforwardly with Rosetta if the bits of the HIV protein are contiguous along the sequence, but it is much harder if the antibody recognizes multiple bits close in three dimensions but far in sequence. In this paper we show how such "discontinuous" epitipes can be transferred from HIV gp120 to a simple scaffold protein. More work will be required to determine whether this or other vaccine candidates designed using this approach will be effective as HIV vaccines-let us all hope so!!
с помощью Розетти хотят разработать такіе белкі, которие можно будет легко проізвесті в огромном колічестве. Анализируются белки части которых присоединяются к белку-цели в какой-то маленькой площади, а сами (эти части) при этом находятся далеко в белковой последовательности
Автор: corsar83 Oct 30 2011, 13:23
Oct 26, 2011
David Baker receives UW Medicine's Inventor of the Year award.
From the award announcement “ the Inventor of the Year award is given to individuals who have translated research from the bench, through partnerships with the biomedical industry, to a product or process that has had a major impact on healthcare and the local economy” . Read more...
Oct 26, 2011
Rosetta@Home software updated to version 3.17
Автор: Rilian Jan 29 2012, 22:38
2012-01-29: Journal post from David BakerLast year we described in Science magazine the design of a new enzyme which catalyzes a chemical reaction called the Diels Alder reaction involving the formation of two carbon-carbon bonds.
This reaction is interesting because no natural enzymes are known to catalyze the reaction. However, it wasn't a very good enzyme, and we asked FoldIt players to try to improve it. As described in Nature Biotechnology this month, remarkably FoldIt players were able to make the designed enzyme 20 times faster by inserting a completely new loop which helps the enzyme bind the chemicals it links together. The combination of Rosetta@Home and FoldIt is turning out to be powerful indeed for solving challenging problems in biomedicine!
Автор: Dromage Mar 12 2012, 10:51
А действительно выгодно и правильно выставлять расчетное время в 1 день? Кто может внятно обьяснить? А то я пока что 6 часов поставил вместо 3-х...
Автор: Rilian Mar 12 2012, 10:57
А действительно выгодно и правильно выставлять расчетное время в 1 день? Кто может внятно обьяснить? А то я пока что 6 часов поставил вместо 3-х...
Каждый таск реально считается например 100000 часов.
то есть от того что твой комп посчитает 6 а не 24 часа в общем проекту не важно
когда я считал только розетту, я ставил 24, просто потому что тратится меньше времени на перезагрузку заданий (ибо в 24:00 оно обрывает и удаляет текущее вычисление даже если оно почти завершено) - таски делятся на кусочки по 10-20 минут, и при обрыве часть такого куска просто пропадает
Автор: Dromage Mar 12 2012, 11:16
Ну а у меня от двухьядерного компа отдано 40% на ВЦГ, 40% на розетту. и 20% на Сетихоум. Какое значение лучше всего МНЕ дла розетты ставить? А то я не очень понял прошлый пример ))
Автор: Rilian Mar 12 2012, 12:57
Dromage, для проекта важнее 24-часовые задания, особенно если ты их оставляешь в памяти при остановке
если ты не оставляешь в памяти либо выключаешь комп каждый день, то 6 часов покатит
Автор: corsar83 Mar 16 2012, 12:04
Mar 14, 2012
Rosetta@Home software updated to version 3.24. This update includes support for symmetry in the hybrid protocol for comparative modeling.
Автор: Death Mar 16 2012, 21:22
тодо добавить в шапко фридс стат
Автор: corsar83 Mar 30 2012, 13:20
Mar 30, 2012
Journal post from David Baker
In the last two months we believe we have made quite a breakthrough in structure prediction, and are excited to test the new method in CASP10. We need your help though--we are now testing many aspects of the new approach and are seriously limited by available CPU cycles. There are now so many flu inhibitor design and structure prediction jobs queued up on Rosetta@Home that there is an eight day wait before they are getting sent out to you. This would be a great time to temporarily increase Rosetta@Home's share on your computers and/or recruit new users--we need all the help we can get!
Автор: Rilian Mar 30 2012, 14:00
Поможем розетте! Подключился
Автор: Rilian Mar 31 2012, 10:46
Прі начале работи нужно загрузіть около 150МБ всякіх файлов
Автор: wolka Mar 31 2012, 10:59
Прі начале работи нужно загрузіть около 150МБ всякіх файлов
У меня на коком-то из ноутов розетта считается, что-то нужно делать?
Автор: Rilian Mar 31 2012, 11:23
Прі начале работи нужно загрузіть около 150МБ всякіх файлов
У меня на коком-то из ноутов розетта считается, что-то нужно делать?
нет, все ок!
Автор: corsar83 Apr 6 2012, 12:48
Apr 5, 2012
Rosetta@Home software updated to version 3.26. This update includes several enhancements for symmetry in the hybrid protocol for comparative modeling.
Автор: ander Apr 8 2012, 10:09
Mar 30, 2012
Journal post from David Baker
...
В останні два місяці, як ми вважаємо, ми зробили певний прорив у передбаченні структури протеїнів і раді протестувати новий метод CASP10. Втім нам потрібна ваша допомога бо ми в даний час тестуємо багато аспектів нового підходу і серйозно обмежені доступними обчислювальними ресурсами. Зараз є так багато структурних дизайнів інгібіторів грипу і робочих завдань у черзі на Rosetta@Home, що наразі восьмий день очікування перед тим як вони будуть отримані вами. Це є певно прекрасний час щоб тимчасово збільшити частку Rosetta@home на комп'ютерах та / або залучити нових користувачів - нам потрібна вся допомога яку можна отримати! Дякую! Девід
Автор: corsar83 Apr 9 2012, 14:05
Apr 8, 2012
Journal post from David Baker
I've described in the past our work using Rosetta and Rosetta@Home to create new enzyme catalysts. In Nature Chemical Biology last month we describe the design of an enzyme which destroys organophosphate
nerve agents and pesticides. These compounds kill by blocking key enzymes, and our designed enzyme eliminates this toxicity. This illustrates how Rosetta@Home enzyme design work can help to solve current problems, including man-made problems.
Автор: Rilian Apr 16 2012, 17:22
Rosetta@Home: Project News Apr 8, 2012
2012-04-08: Journal post from David BakerI've described in the past our work using Rosetta and Rosetta@Home to create new enzyme catalysts. In Nature Chemical Biology last month we describe the design of an enzyme which destroys organophosphate nerve agents and pesticides. These compounds kill by blocking key enzymes, and our designed enzyme eliminates this toxicity. This illustrates how Rosetta@Home enzyme design work can help to solve current problems, including man-made problems.
Автор: Rilian Apr 16 2012, 22:24
Хмм, а чекпоинтов тут и раньше не было ? о_О
Автор: corsar83 Apr 17 2012, 09:10
Чекпоинты есть и были. Может последняя версия боинка глючит.
Автор: Rilian Apr 28 2012, 13:11
проект участвует в http://distributed.org.ua/forum/index.php?showtopic=5602 с 5 по 10 мая.
Все очки полученные с 5 по 10 мая включительно идут в зачет
если у вас есть возможность, приготовьте мега-слив ![]()
Автор: Rilian Apr 28 2012, 16:56
Rosetta@Home: Project News Apr 27, 2012
2012-04-27: Rosetta@Home software updated to version 3.30. Addresses issues with Cartesian Relax and the modeling of disulfides in the hybrid protocol for comparative modeling.
Автор: Rilian Apr 29 2012, 05:04
http://boinc.bakerlab.org/rosetta/rah_old_potd.php
Feb 03, 2012
Predictor of the day: Congratulations to ramok (Team Ukraine) for predicting the lowest energy structure for workunit ProteinG_abinitio_SAVE_ALL_OUT_design_relax_g009_007_25077_0 !
Mar 30, 2010
Predictor of the day: Congratulations to Real_penguine (Team Ukraine) for predicting the lowest energy structure for workunit lrmix_seq_score12_mix_rlbn_1faa__18593_0 !
Mar 08, 2010
Predictor of the day: Congratulations to Piligrimus (Team Ukraine) for predicting the lowest energy structure for workunit lr_mix_sc12_icoor_run01_A_rlbd_1h75___16605_0 !
Dec 29, 2009
Predictor of the day: Congratulations to abishai (Team Ukraine) for predicting the lowest energy structure for workunit resa_sel_core_1.5_low200_beta_low200_nostart_hb_t374___16310_0 !
Nov 27, 2009
Predictor of the day: Congratulations to Administrator (Team Ukraine) for predicting the lowest energy structure for workunit lr5_dun08_it02_B_rlbd_1o5u_SAVE_ALL_OUT__15457_0 !
Nov 04, 2009
Predictor of the day: Congratulations to giggs (Team Ukraine) for predicting the lowest energy structure for workunit lr5_combine_smooth_torsion_it02_A_rlbn_1poh_SAVE_ALL_OUT__14883_0 !
Sep 12, 2009
Predictor of the day: Congratulations to smudgy (Team Ukraine) for predicting the lowest energy structure for workunit lr8_newhb_run02_rlbd_1a19__14605_0 !
Jul 11, 2009
Predictor of the day: Congratulations to uNiUs (Team Ukraine) for predicting the lowest energy structure for workunit lr5_D_rama_map_iter05_notsq_rlbn_2vik_SAVE_ALL_OUT_NATIVE_NOCON_12680_0 !
Jul 14, 2008
Predictor of the day: Congratulations to Admin (Team Ukraine) for predicting the lowest energy structure for workunit FR_298_relax_3292_0 !
Apr 10, 2007
Predictor of the day: Congratulations to Toxa (Team Ukraine) for predicting the lowest energy structure for workunit BENCH_02JUMPING_SAVE_ALL_OUT_1iibA_NATIVE_PAIRS_SEQSEP5_TRUESS_OEG_filters_1567_0 !
Dec 25, 2006
Predictor of the day: Congratulations to Alexey (Team Ukraine) for predicting the lowest energy structure for workunit s014__BOINC_ABRELAX_SAVE_ALL_OUT_hom007__1410_0.clean.sc !
Автор: Rilian Apr 30 2012, 13:47
Rosetta@Home: Project News Apr 29, 2012
2012-04-29: Journal post from David Baker
I have just been told the very good news that Rosetta@home will be the first project of the BOINC pentathlon, and would like to thank all of the participating teams. I also just learned from the discussion thread that Rosetta@home will be the project of the month for BOINC synergy-this is more excellent news!!Your increased contributions to rosetta@home could not come at a better time! We've been testing our improved structure prediction methodology in a recently started challenge called CAMEO. For most of the targets, the Rosetta@home models are extremely good, but for a minority of targets the predictions are not good at all. We've now tracked down the source of these failures and it is what we are calling "workunit starvation"; in the limited amount of time the Rosetta server has to produce models (2-3 days) in these cases very few models were made-this happens because many targets are being run on the server so that only a fraction of your cpu power is focused on any one target. while we are working to fix this internally, by far the best solution is to have more total CPU throughput so each target gets more models. You can follow how we are doing at http://www.cameo3d.org/. You will see that Robetta is one of the few servers whose name is not kept secret-this is because Rosetta is a public project. Our server receives targets from CAMEO and soon CASP, sends the required calculations out to your computers through Rosetta@home, and then processes the returned results and submits the lowest energy models. We are excited that the workunit starvation problem may go away through your increased efforts for Rosetta@home. Thanks!!!
ітак, Розетта участвует одновременно в двух соревнованіях по моделірованію белков! ![]()
http://www.cameo3d.org/modeling/index.html
Автор: Rilian May 7 2012, 16:10
Pentathlon рулит ![]()

Автор: Khvastov Maxim May 7 2012, 16:58
Rilian, думаешь с 10-го числа такой пик будет на графике команды в WCG? ![]()
Автор: Rilian May 10 2012, 13:16
Кстати поднялись на 40 место http://boincstats.com/stats/team_stats.php?pr=rosetta&st=0#40
Автор: Rilian Jun 10 2012, 22:14
новости из журнала Девида Бейкера
Many common materials such as silk and wool are made out of regular repeating arrays of proteins, and symmetric protein arrays make up the coats of viruses and many other assemblies inside cells. The ability to robustly design self assembling materials made out of proteins would have huge numbers of applications-the naturally occurring assemblies are useful, but imagine if we could make custom materials for 21st century problems. In this weeks Science magazine, we describe the use of Rosetta to design self assembling protein nano structures with very high accuracy. We are now attempting to create many different types of symmetric materials, and you will be seeing more symmetric calculations on your rosetta@home screen server. Thank you for making possible this completely new approach to nanotechnology!
Автор: Bel Aug 13 2012, 21:28
Кусок поста из форума TSC! Russia... обсуждение CASP 10.
Так CASP10 закончился? Начался нормальный просчет новых молекул, а не чемпионат, кто быстрее предскажет уже известное?
Мне сейчас идут в основном требующие мало памяти задания типа "Tc734_ab_relax_cst_t000___dekim_08_08". Что это приблизительно может считаться?
Еще не закончился, но подоходит к концу. Крайний срок сдачи последних предсказаний - 18 августа:
http://predictioncenter.org/casp10/targetlist.cgi (кстати по этой ссылке - по тем белкам у которых срок сдачи прошел в последнем столбце можно найти ссылки на подробную информацию с описанием что это был за белок)
А в R@H по-моему уже дейсвительно считать CASP перестали, тем что осталось robetta сервера занимаются + ручная обработка.
Небольшая статистика с CASP10.
Все на соревнование было выставлено 114 белков с полностью неизвестной структурой(+43 цели, где нужно было работать по частично известным данным). По ним получено 65000 предсказаний - каждая команда может несколько(если правильно помню 5) лучших своих моделей прислать по каждой цели.
Причем не совсем правильно говорить, что это еще раз перемалывают "старое и известное". Особенность CASP в том, что считаются белки стуктура которых была опеределена (экспериментально) совсем недавно(от дней до 1-2 года) и еще нигде не была опубликована. Т.е. в отличии от обычных "тренировок" и тестирований алгоритмов поиск идет полностью в слепую(двойной слепой метод). А оценивает все это независимая(от участников) команда. Что позволит достоверно сказать кто есть ху? На данный момент. Так что это не просто "чемпионат", а и метод оценки эффективности работы разных научных групп. Который может влиять в т.ч. на распределие грантов, пожертвований и т.д. в т.ч. поддержку РВ в у тех групп, которые РВ используют (а то каждый хвалит себя конечно в первую очередь, а как не будучи специалистом понять, где серьезная и эффективная наука, а где больше пиара? например Бейкер заявляет, что по их собственной оценке за последние 2 года они продвинулись в качестве моделирования больше, чем за предыдущие 6 лет - посмотрим как это отразится на результатах независимой проверки)
Кстати проме собственно предсказания структуры там есть и другие категории "соревнования", например поиск активных центров белка (так же только по линейной аминокислотной формуле)
Да, отыскал результаты с предыдущего CASP9( http://predictioncenter.org/casp9/groups_analysis.cgi )
В категории скоростной(результат возвращался в среднем через 2.4 суток после получения задания при предельно допустимых 3) автоматизированный поиск структуры Розетта тогда заняла 11е место (из 79 участвовавших команд).
В открытой смешанной категории (как полность автоматизированный так и машина+человек на кот. дается гораздо больше времени) лаба бейкер заняла
36е (полный авто) и 29е (с участием человека) места из 174 участвовавших групп
Для сравнения например POEM занял тогда 119/174 место (и в условных попугаях результаты где-то в 3 раза слабее/хуже чем у лабы Бейкера).
F@H как понимаю вообще не участвовал.
Так что осенью(когда будут подведены результаты CASP10) посмотрим кто как продвинулся за прошедшие 2 года.
Автор: Rilian Aug 13 2012, 21:31
Очень познавательно![]() !!!
!!!
А кто занял 1 место? на чем они считают и если у них такие крутые алгоритмы, то почему все их не используют ?
Автор: Bel Aug 13 2012, 21:49
Rilian, я сам в этом разбираюсь на уровне раздупления новостей и прочей информации. ![]()
Пока что соревнование идет. Нужно ждать новостей на форуме розетты, через какое то время.
Кстати POEM@Home тоже принимает участие в этом году. Правда только на цпу, поэтому может и не смогут обработать большой объем информации.
Автор: ander Oct 5 2012, 14:05
Очень познавательно
А кто занял 1 место? на чем они считают и если у них такие крутые алгоритмы, то почему все их не используют ?
WCG HPF - це по суті і є Rosetta@Home, але Rosetta@Home - дослідницький проект, у якого дві мети: дослідження обчислювальних методів та винайдення нових структур.
Тому Rosetta@Home
1) має код клієнта менш оптимізований під нові процесори та відеокарти ніж інші проекти
2) має наукову перевагу над іншими проектами розподілених обчислень бо вдосконалює свої методи, що дозволяє rosetta@home показувати непогані результати у CASP (серед дослідницьких проектів).
Both Phase I and Phase II of the Human Proteome Folding Project (HPF), a subproject of World Community Grid, have used the Rosetta program to make structural and functional annotations of various genomes. Although he now uses it to create databases for biologists, Richard Bonneau, head scientist of the Human Proteome Folding Project, was active in the original development of Rosetta at David Baker's laboratory while obtaining his PhD. http://en.wikipedia.org/wiki/Rosetta@home
Автор: Александр Oct 22 2012, 20:53
Новичок, не знаю как это сделать. Был перерыв 3 года. До этого в команде не состоял.
Автор: Rilian Oct 22 2012, 23:06
Новичок, не знаю как это сделать. Был перерыв 3 года. До этого в команде не состоял.
привет
в каком проекте?
Автор: Александр Oct 23 2012, 12:25
Rosetta. Только хотел подключиться к Rosetta@home, но получилось mini.
Автор: Rilian Oct 23 2012, 12:31
Rosetta. Только хотел подключиться к Rosetta@home, но получилось mini.
ок, это немного не относится к форуму Змагання. ?
в розетте ядро рассчетов называется rosetta mini. все ок
если будут вопросы по проекту, пиши в соответствующем форуме http://distributed.org.ua/forum/index.php?showforum=26
как подключиться, тут есть полная инструкция http://distributed.org.ua/forum/index.php?showtopic=865
Автор: Александр Oct 27 2012, 10:01
Спасибо.
Автор: corsar83 Nov 10 2012, 11:25
Nov 8, 2012
Journal post from David Baker
With your help, we have made an exciting breakthrough in protein design that is reported in a research article titled "Principles for designing ideal protein structures" in the journal Nature today. You can read about it at
http://www.nature.com/news/proteins-made-to-order-1.11767
In this paper, we describe general principles for creating new proteins from scratch. The new Institute for Protein Design is using these principles to design new proteins to treat disease.
Rosetta@home was absolutely critical to this work as described in the news article; Figure 3 in the paper shows how all of your contributions were used to test designed sequences to see if they folded up to the right structure. Most of the work units we are sending out on Rosetta@home these days are for exactly these kind of tests on the new proteins we are designing--this is absolutely critical to the research and to the development of new therapeutic and other functions. Thank you again for all of your contributions!
Автор: Alien Nov 10 2012, 17:43
крутая новость... жду когда интегрируют алгоритмы в другие программы)
WCG HPF - це по суті і є Rosetta@Home, але Rosetta@Home - дослідницький проект, у якого дві мети: дослідження обчислювальних методів та винайдення нових структур.
Тому Rosetta@Home
1) має код клієнта менш оптимізований під нові процесори та відеокарти ніж інші проекти
2) має наукову перевагу над іншими проектами розподілених обчислень бо вдосконалює свої методи, що дозволяє rosetta@home показувати непогані результати у CASP (серед дослідницьких проектів).
Наверное, корректней сказать поиск нативной конформации по а.п.=)
Автор: Bel Nov 10 2012, 20:22
Публикация точно лабы Бейкера. (Вот упрощенная журналистская версия, там же подкаст есть в исполнении самого др.Бейкера: http://www.nature.com/news/proteins-made-to-order-1.11767)
И одна из прорывных и важных за последнее время.
А вы сэр громко сели в лужу со своими высказываниями типа
> вся проблема в том, что уж слишком давненько они обкатываются.
Давненько обкатывается решение задачи заключающейся в том, чтобы по известной первичной структуре (аминокислотной последовательности, которые уже известны для всех белков человека и многих животных) определить третичную структуру (3д модель свернутого белка).
Этой задачей ученые из множества лабораторий и университетов действительно уже больше десятка лет занимаются. Ну так вот - на данный момент она уже фактически решается для достаточно сложных белков (примерно до 600-1000 аминокислот, что перекрывает большинство натуральных белков кроме самых больших и сложных). И один из самых главных вкладов в этот прогресс внесла как раз Розетта и в частности R@H . Последние масштабные испытания ее алгоритмов показывают, что для большинства белков эта задача уже решается за разумное время (от нескольких дней до нескольких недель - при условии использования для расчетов суперкомпьютера или распределенной вычислительной сети) и с приемлемой (для практического использования) точностью.
А в этой последней статье речь идет уже об обратной задаче. На основе придуманной 3д структуру белка (а значит и его функций и свойств) определить его первичную структуру (аминокислотную формулу). Т.к. зная эту формулу, синтезировать нужный белок это уже легкая (по современным меркам) задача.
К решению этой задачи (в общем виде - для произвольного белка) сейчас еще только приступают, и этот результат один из первых заметных успехов на этом пути. И кстати белки по 70-100 аминокислот(1-2 тысячи атомов) с которыми работали не совсем уж такие маленькие - в некоторых случаях этого уже может быть достаточно для практического применения. И сомневаюсь, что формулу для таких белков можно "подобрать вручную". Вот один из созданных белков: http://www.rcsb.org/pdb/explore.do?structureId=2kl8
А между тем, для разработки лекарств (а так же создания искусственных организмов с запрограммированными свойствами) эта задача даже важнее 1й. Т.к. обычно выяснить структуру целевого белка (например, критически важного белка в вирусе или болезнетворной бактерии) уже не представляет особых сложностей (а зачастую они и так уже известны - определены экспериментально с использованием рентгеновской кристаллографии и/или ядерно-магнитного резонанса). Определить в нем "активные центры" (воздействием на которые можно заблокировать/изменить функцию этого белка) тоже уже не очень сложно. Проблема заключается в том, чтобы найти (или создать) другой белок, который бы нужным образом взаимодействовал с этим(и) активным центром целевого белка. Сейчас для этого пытаются применять(не очень успешно) методы тупого перебора (моделирования взаимодействия) огромного числа случайных кандидатов + "направленная эволюция" после того (точнее если) нашелся хоть один кандидат, хотя бы с минимальной желаемой активностью (в него вносятся случайные мутации - замены 1-2 аминокислот, моделируют взаимодействие цель-кандидат заново, смотрят слабее или сильнее оно стало, делают следующую замену и т.д.).
После же достаточного развития методов изложенных в этой работе, для разработки белка-лекарства (или белка-«механизма») можно будет не тыкаться перебором, а просто рассчитать какая у белка должна быть аминокислотная формула, чтобы при ее сворачиванни сформировалась структура взаимодействующая с активным центром белка-мишени. И имея аминокислотную формулу - его легко синтезировать и проверить экспериментально.
ну а уж от того, что сейчас есть до "The new Institute for Protein Design is using these principles to design new proteins to treat disease." такая пропасть, что даже представить ее тяжело на самом деле
А тут еще один жирный плюх. Такой институт до которого "такая пропасть" УЖЕ как раз создан совсем недавно и реальная работа по описываемой схеме уже началась! И др.Бейкер станет главной этого нового института. См. предыдущие новости на главной проекта, а подробности тут : http://depts.washington.edu/ipd/
(Еще была большая статья с виртуальной экскурсией по новому институту, но пока не было времени перевод сделать)
P.S.
Кстати в честь официального открытия этого нового института, грозятся отобрать несколько кранчеров (5-10 человек) из активных в R@H и пригласить уже на реальную экскурсию туда.
Добавлено спустя 44 минуты 13 секунд:
Еще поробности относительно статьи. Опубликовали имена кранчеров, кому повезло и досталась жаба в которой была определена наилучшая структура белка (из 5 описываемых в статье):
Fold-I : Aalelan (United States)
Fold-II : Jef (United States)
Fold-III : georgebg (Bulgaria)
Fold-IV: medvjet009(Czech Republic)
Fold-V : _2e_ (Russia).
------------------------------------------------------
Пост взят из форума команды TSC! Russia.
Автор: Bel Dec 24 2012, 12:44
Результаты CASP 10!
Hi ALL ROSETTA@HOME participants,
Good new from CASP10 meeting, which just ended yesterday at Italy!!! Having your contribution to Rosetta@Home project, the Baker lab did really good this time. With a lot of exceptional scientists' effort, in CASP10 we have improved our template-based modeling (TBM) method. And here are those results:
Let's focus on TBM easy/hard using "best model" (best out of five submission) as the criterion.
1. Baker Lab is ranked first in Server Prediction (fully automated structure prediction)
---------------------------------------------------
[1] [2] [3] [4] [5]
---------------------------------------------------
1. 330 s BAKER-ROSETTASERVER 111 79.614
2. 035 s Zhang-Server 111 79.365
[1] rank
[2] server id
[3] server name
[4] proteins count
[5] Z-score (how well you did compared to others using GDT-TS as metrics)
website: http://www.predictioncenter.org/casp10/groups_analysis_best.cgi?type=server&tbm=on&tbm_hard=on&submit=Filter
2. Baker Lab is ranked second in Human Prediction (manually structure prediction):
---------------------------------
[1] [2] [3] [4] [5]
---------------------------------
1. 237 zhang 56 56.832
2. 477 BAKER 56 54.482
[1] rank
[2] server id
[3] server name
[4] proteins count
[5] Z-score (how well you did compared to others using GDT-TS as metrics)
website: http://www.predictioncenter.org/casp10/groups_analysis_best.cgi?type=all&tbm=on&tbm_hard=on&submit=Filter
3. Baker Lab is ranked first in Human Contact-assisted Prediction (given few native contacts, to predict protein structure) with a tremendous distance to the group ranked second:
Here is the preliminary results the CASP organizer posted on website:
http://predictioncenter.org/casp10/results.cgi?view=targets&model=all&tr_type=others&groups_id=4
, where for each graph with orange-color lines into it, you need to compare the black line to the rest- that's us. The main predictor, David Kim, will come here, explaining it clearly to you all with a summary table.
With the help and contribution to Rosetta@HOME from you all, we are able to make significantly progress to predict more accurate protein structures, which ends up would impact the real world by tackling human deseases, like, therapeutics protein design.
Please let me know if I said something unclear. Again, thank you so much for the support. We will keep you updated about our triumph in contact-assisted protein structure prediction in CASP10.
Sincerely,
Baker Lab CASP10 Team
-----------------------------------------------------------------
Hi all,
As Ray posted below, we did well! Here are a couple plots and images of our results from the contact assisted category. Points above the line are best. The y-axis is the GDT (how close we are to the correct structure, higher is better) of our best models for each of the contact assisted targets vs on the x-axis for the left plot, the GDT of the best models of all predictions without using contact information, and for the right plot, the best models of other predictors using contact information. As you can see we are above the line for many of the targets. An outstanding prediction was for T0680o, which is a tetramer. There were other outstanding predictions. See below.
Thank you all for contributing to this effort. I'll see if I can find users who provided the actual predictions that led to the final submitted models. It might be tough though.





Автор: Rilian Dec 24 2012, 13:04
Great success!
Автор: Rilian Mar 11 2013, 15:37
Mar 11, 2013
http://boinc.bakerlab.org/rosetta/forum_thread.php?id=1177&nowrap=true#75215
We have learned to design a new class of proteins which could be useful both as drugs and in sensors. As I've explained before, most drugs are small molecules with fewer than 50 atoms. There are many drugs, such as blood thinning agents, which are very dangerous if given in too large doses. We have succeeded in designing proteins which bind to specific small molecules very tightly. These proteins could be used as antidotes in case of overdose with the target small molecule-for example we've designed a protein which could be useful to treat toxicity due to overdose of the drug digoxin used to treat heart disease. With collaborators, we are working to use these designed proteins to detect levels of the target small molecules in the blood or in the environment.
Автор: eugeny Apr 27 2013, 06:27
Приложение http://boinc.bakerlab.org/rosetta/apps.php обновили до версии 3.46.
Автор: Rilian May 23 2013, 16:43
David Baker's Rosetta@home journal
http://boinc.bakerlab.org/rosetta/forum_thread.php?id=1177
Posted 20 May 2013
We have discovered how to make several new classes of protein structures! Rosetta@home has been absolutely critical in this work: when we design a sequence to fold into a new structure, the last thing we do before ordering a synthetic gene so we can make the protein in the laboratory is to send it out to you to predict the structure-if it folds to the structure we designed, we go ahead with it, but if you find that the lowest energy state is a different structure we go back to the drawing board. Our success rate in making brand new structures is far higher than I or anybody else ever expected, and the reason the success rate is so high is that your calculations provide a very stringent test of whether the designed sequence will actually fold the way it is supposed to. In the next few weeks I and other scientists here will describe to you the new classes of proteins we are making, and the many applications they will be useful for. Thank you for your absolutely essential contributions to this newly emerging scientific field!
-- David Baker
Автор: Rilian Jun 24 2013, 14:28
To our awesome Rosetta@Home contributors:
At the beginning of 2012 we told you that it looked like eight designed proteins stuck to EED as desired. Further experiments showed us that of our eight initial hits, only two were real, and those two proteins were too weak to be improved with laboratory techniques. We've gone back to the drawing board, using more aggressive modeling techniques to design better binding proteins to mimic Ezh2. It turns out that no known protein structure is similar enough to the Ezh2 binding helix to allow us to simply transplant key amino acids from Ezh2 to a new host protein, as we tried before. What were doing now is making new proteins from scratch that have the exact shape that we need to mimic Ezh2. The "EED" runs you'll now see on Rosetta@Home are structure prediction of designs to see if they have the desired curvature when Rosetta folds them up. Thanks again for all of your donated computer time!
For those new to this thread/message board, here is what I wrote previously about the EED project:
The second of these interactions involve a protein called EED and another called Ezh2. If you think about DNA as a ladder, then each of our cells has about six billion rungs worth of ladder. In order to keep all of that DNA from getting hopelessly tangled, our cells keep it on little spools, called histones, when not in use. Think of this like a massive film archive. It turns out that there is a lot of DNA that never gets used, so the cells put special flags on those spools (histones) that say something like "never use this section of DNA". EED is a large protein that attaches to those flags and brings along Ezh2, a "flagging machine" for the ride. EED makes sure that Ezh2 adds flags to histones that are supposed to get them, and not to histones carrying DNA the cell actually wants to use. Some cancer cells reduce the amounts of EED or Ezh2 to prevent flagging of DNA regions that they want to use to take over our bodies. Other cancer cells expand the amounts of EED or Ezh2 to flag DNA regions that are getting in the way. Scientists are working hard to better understand how EED, Ezh2, and friends work in normal cells and what goes wrong in cancer cells. Some are trying to develop drugs that prevent Ezh2 from attaching to EED. This means that the drugs have to stick to EED more tightly than Ezh2. Although Ezh2 attaches relatively loosely to a large patch on the surface of EED, their aren't any drugs yet available that do what we want.
I'm trying to make proteins that will do the same job. I used Rosetta@home to design a set of proteins that mimic Ezh2 in order to block it from attaching to EED. Just to give you a sense of how much computing I need for a project like this, I submitted just under two million work units to Rosetta@home, and donor's computers ran my protocol just under a billion times to give me about 54 designs to look at, from which fourteen were suitable for testing. This took about a month to run on Rosetta@home. From initial experiments, it looks like eight of the designs stick to EED, and one in particular sticks three times better than the Ezh2 found in our cells. Now I'm working to improve the best design at the lab bench and hope to send it to co-workers for testing in living cells. There's still a lot of work to be done to make sure that everything is working right with this design, but I want to give a big thank-you to all of you who donated your computer time to make this possible! For more information about EED, Ezh2, and related proteins, please visit:
http://en.wikipedia.org/wiki/EED
http://en.wikipedia.org/wiki/SUZ12
http://en.wikipedia.org/wiki/EZH2
http://en.wikipedia.org/wiki/PRC2
http://en.wikipedia.org/wiki/Polycomb-group_proteins
Work units for this project carried the word "EED" in their name.
http://boinc.bakerlab.org/rosetta/forum_thread.php?id=4477&nowrap=true#75771
![]()
![]()
![]()
![]()
![]()
Автор: Rilian Sep 15 2013, 13:22
Набрав 500К ![]()
![]()
Автор: CrazyBigCat May 7 2014, 10:39
Rosetta@home включен в дисциплину Natural Sciences в 5th BOINC Pentathlon 2014 - любители медицинских проектов подключайтесь![]() Еще есть время "нахомячить" заданий и помочь команде UKRAINE в соревнованиях...
Еще есть время "нахомячить" заданий и помочь команде UKRAINE в соревнованиях...
Автор: Asket May 9 2014, 21:34
Парни, а зачем "Для большей научной пользы выставьте время рассчета ВЮ - 1 день.", что конкретно это дает? Обьясните пожалуйста.
Автор: Rilian May 9 2014, 21:39
Asket, розетта рандомно подбирает конфигурацию белка. Теоретически выставив самое больше время попытки каждая структура будет исследована максимально возможным кол-вом способов. То есть вдруг именно в самом конце работы ВЮ комп найдет самую классную конфигурацию ?
А вообще, в данный момент и еще 2 дня будет идти BOINC Pentathlon http://distributed.org.ua/forum/index.php?showtopic=6361 , и в нем очень рекомендуется выставить время ВЮ на 1 час, для максимальной отдачи http://boinc.bakerlab.org/rosetta/prefs.php?subset=project
Автор: Asket May 10 2014, 11:12
Rilian, понял, спасибо.
Автор: Arbalet Jan 9 2015, 17:31
http://boinc.bakerlab.org/rosetta/forum_thread.php?id=6574&nowrap=true#77808 краткие результаты эксперимента CASP11, считают его успешным, благодарят участников за неоценимую помощь в исследованиях и поздравляют с Новым Годом.
Автор: Arbalet Mar 24 2015, 23:25
Поделились http://bcove.me/860jiohx (руководитель проекта) на съезде Американской ассоциации содействия развитию науки (American Association for the Advancement of Science - AAAS), где он рассказывает об исследованиях в своей лаборатории и в проекте.
Автор: Arbalet Sep 8 2016, 00:27
А вы вообще в курсе, что в Rosetta@home достигли больших результатов в проектировании белковых структур? Говорят чуть ли не о революции в фолдинге белков! Обложка журнала Science во весь разворот посвящена этому событию ![]() Надо переводом заняться
Надо переводом заняться
Автор: Rilian Sep 14 2016, 23:31
Не в курсе) Присылай ссылки на любом языке!
Автор: langolierra Nov 3 2016, 12:01
Пишуть, що программа достатньо точно обчислює лише малі білки у яких менше 100 амінокислот в ланцюжку. З більшими не все так просто і засновник, наскільки я зрозумів, сумнівається чи взагалі можливо . http://www.sciencemag.org/news/2016/07/protein-designer-aims-revolutionize-medicines-and-materials
Автор: gladiator_maximus Mar 23 2020, 08:37
Роль Розетты в борьбе с коронавирусом
https://www.ipd.uw.edu/2020/02/rosettas-role-in-fighting-coronavirus/
Автор: ale4316 Mar 26 2020, 11:25
Задания по короновирусу составляют процентов 70-80 от общих заданий. Памяти берет около 1,5 гиг на задание.
Автор: dimus8210 Mar 26 2020, 13:55
Задания по короновирусу составляют процентов 70-80 от общих заданий. Памяти берет около 1,5 гиг на задание.
не звернув увагу що мізків жере 1.5Гб, в результаті повісило систему наглухо вижравши всі 16Гб при 16 потоках, довелось виставити 50% цп. на 2-х xeon 2680 боюсь запускати бо озу 12Гб, будуть ядра простоювати. треба розкошелитись хоча б на 32Гб, блін
Автор: Arbalet Mar 26 2020, 14:20
Задания по короновирусу составляют процентов 70-80 от общих заданий. Памяти берет около 1,5 гиг на задание.
Переключаю ЦПУ на Розетку, все равно F@h какие-то левые проекты в основном гонит на проце.
Автор: 5erg Mar 28 2020, 09:21
Задания по короновирусу составляют процентов 70-80 от общих заданий. Памяти берет около 1,5 гиг на задание.
не звернув увагу що мізків жере 1.5Гб, в результаті повісило систему наглухо вижравши всі 16Гб при 16 потоках, довелось виставити 50% цп. на 2-х xeon 2680 боюсь запускати бо озу 12Гб, будуть ядра простоювати. треба розкошелитись хоча б на 32Гб, блін
Давай я с оперативкой помогу? drop_table_password в телеге
Автор: Arbalet Apr 3 2020, 13:40
Розетта здорового человека:
Автор: gladiator_maximus Apr 3 2020, 13:55
В связи с недавней вспышкой COVID-19 R@h использовался для прогнозирования структуры белков, важных для заболевания, а также для производства новых стабильных мини-белков, которые можно использовать в качестве потенциальной терапии и диагностики, таких как показанный выше, который связан с частью белка шипа COVID-19.
Чтобы помочь нашим исследованиям, мы рады объявить о новом обновлении приложения, и благодаря помощи сообщества разработчиков Arm, включая Рекса Сент-Джона, Дмитрия Москальчука, Дэвида Тишлера, Ллойда Уоттса и Сахаджарупа, мы рады также включить платформу Linux-ARM. В этом обновлении мы продолжим делать белковые связующие для COVID-19 и связанных с ними задания с использованием новейшего источника Rosetta.
Спасибо волонтерам R@h за постоянную поддержку этого проекта. Часы вашего процессора используются не только для точного моделирования структур важных белков, но и для разработки новых. Давайте объединимся и сразимся с COVID-19!
Основным способом взаимодействия белков друг с другом является прилипание друг к другу. Как вы могли видеть из графического приложения R@h, белки бывают разных форм и размеров. По этой причине большинство белков не прилипают случайным образом друг к другу, а скорее прилипают очень специфично к горстке других белков. Например, вирусный спайковый белок COVID-19 прилипает к человеческому белку ACE2, благодаря которому вирус попадает в клетку.
IPD усердно работает над улучшением способности создавать такие связывающие взаимодействия. Этот процесс начинается с создания набора каркасных белков, которые не имеют иной цели, кроме точного сворачивания в атомную структуру. Эти строительные леса затем закрепляются на целевом белке, представляющем интерес, и их поверхности предназначены для идеального дополнения цели. Наконец, проекты оцениваются, фильтруются и тестируются на связывание в лаборатории.
Теперь мы будем использовать R@h, чтобы выполнить этап проектирования поверхности. Стыковка и фильтрация выполняются быстро, но на самом деле разработка белка происходит медленно. Мы будем использовать огромное количество вычислительной мощности, доступной на R@h, для выборки каждой аминокислоты в каждой позиции на границе раздела. Затем мы выберем лучшие комбинации аминокислот с использованием имитации отжига и Монте-Карло. Выборка является ключевой для этого процесса, и поэтому мы обращаемся к R@h.
Итак, присоединяйтесь к нам в ближайшие недели, когда мы создадим связующие вещества для COVID-19 и родственных белков. Мы все еще будем делать предсказание структуры и проектирование скаффолдов, поскольку они также крайне важны для белковой науки. Но обратите внимание на случаи разработки интерфейса, потому что кто-то может разрабатывать следующее лекарство от COVID-19.
И, надеюсь, вы останетесь, как только пандемия закончится. Мы можем создавать такие связующие только потому, что много лет работали над этой проблемой. Впрочем, впереди еще долгий путь. Совершенствование науки требует времени и вычислений, поэтому мы надеемся, что вы присоединитесь к нам в этой увлекательной поездке.
- Брайан Ковентри
https://boinc.bakerlab.org/rosetta/forum_thread.php?id=13702#93153
Автор: dimus8210 Apr 7 2020, 21:40
вже деякі завдання з'їдають по 2.3Гб оп
Автор: Dmitro Apr 24 2020, 21:45
Были всякие Годзилы в команде, но и Титаны заглядывают.
Лям в сутки
https://boinc.bakerlab.org/rosetta/show_user.php?userid=191322
http://boinc.bakerlab.org/rosetta/team_members.php?teamid=328&offset=0&sort_by=expavg_credit
Автор: dimus8210 Apr 25 2020, 00:27
Были всякие Годзилы в команде, но и Титаны заглядывают.
Лям в сутки
https://boinc.bakerlab.org/rosetta/show_user.php?userid=191322
http://boinc.bakerlab.org/rosetta/team_members.php?teamid=328&offset=0&sort_by=expavg_credit
переглядаючи цей профіль почуваєш себе якоюсь креветкою поряд з синім китом
сподіваюсь розетту включать до пентатлону і Technologov буде з нами
Автор: Dmitro Apr 25 2020, 14:00
переглядаючи цей профіль почуваєш себе якоюсь креветкою поряд з синім китом
сподіваюсь розетту включать до пентатлону і Technologov буде з нами
С другой стороны, неужели всё так плохо с Короновирусом, что подлючают такую "тяжелую артилерию" в лице 8 места по рейтингу.
Автор: ale4316 Apr 30 2020, 05:58
Тссс. Не вспугните. Он нам очень нужен.
Автор: Rilian Aug 8 2024, 06:01
Додаток Rosetta було оновлено, щоб включити виправлення в основний алгоритм для моделювання білка.
Через те, як алгоритм взаємодіє всередині, ми залишимо графіку вимкненою для цієї версії.
Будь ласка, повідомляйте про будь-які проблеми в ланцюжку обговорень тут: https://ralph.bakerlab.org/forum_thread.php?id=944
Автор: Rilian Aug 16 2024, 03:02
Я отримав декілька завдань
Автор: dimus8210 Oct 9 2024, 12:58
Нобелівську премію з хімії присудили за дизайн і прогнозування структури білків. У 2024 році половину премії отримав Девід Бейкер (David Baker) за комп'ютерний дизайн білків, а іншу половину — Деміс Гассабіс (Demis Hassabis) і Джон Джампер (John M. Jumper) за передбачення їхньої структури. Лауреатів оголосили під час онлайн-трансляції на сайті Нобелівської премії
Invision Power Board
© Invision Power Services