Привіт Гість ( Вхід | Реєстрація )
| Dimych |
 May 29 2007, 12:12 May 29 2007, 12:12
Пост
#1
|
|
Так, я створив профіль! Група: New Members Повідомлень: 2 З нами з: 29-May 07 Користувач №: 522 Стать: Чол |
 Проект "Rosetta@home" ---------------------------------------------------------------------------------------------------------- Официальный сайт Официальная статистика по команде "Ukraine" ТОП-20 участников:  ---------------------------------------------------------------------------------------------------------- Дата основания команды - 12.04.2006 Капитан - uNiUs ---------------------------------------------------------------------------------------------------------- Для присоединения к команде Украины: 1. Загрузите BOINC менеджер (Если его у Вас еще нет!) 2. Перейдите в "расширенный вид" 3. Выберите сервис ---> добавить проект 4. Введите адрес проекта http://boinc.bakerlab.org/rosetta 5. Введите свои регистрационные данные. 6. Найдите нашу команду. Она называется Ukraine и адрес ее статистики вы могли видеть выше. 7. Если есть доступные для загрузки задания Вы их получите и начнете расчеты. ---------------------------------------------------------------------------------------------------------- BOINC 6 Versions Change Log ---------------------------------------------------------------------------------------------------------- Полезная информация: Для идентификации пользователя в BOINC могут служить 2 вещи: 1) пара e-mail/пароль 2) межпроектный идентификационный ID (Cross-project ID) - 32значное шестнадцатиричное число. Если Вы пожелаете подключится ещё и к другому BOINC-проекту, то помните: чтобы не плодить новых аккаунтов при подключении к новому проекту или команде, нужно обязательно везде регистрироваться с одним и тем же e-mail/паролем либо CPID. если при регистрации в проекте указать другие e-mail или пароль, BOINC создаст новый аккаунт с тем же именем! ---------------------------------------------------------------------------------------------------------- Если у вас есть желание помочь команде, посчитать на командный аккаунт, можно использовать так называемый "слабый ключ аккаунта" (weak account key) который позволяет коннектиться к проекту не вводя пароли. Все очки будут присваиваться командному юзеру distributed.org.ua Чтобы подключиться: 0) установите BOINC 1) создайте файл account_boinc.bakerlab.org_rosetta.xml в папке \data\ 2) в файл поместите текст CODE <account> <master_url>http://boinc.bakerlab.org/rosetta/</master_url> <authenticator>46ed7286c5974c66fe43ee6b4828413a</authenticator> <project_name>rosetta@home</project_name> </account> 3) перезагрузите BOINC ---------------------------------------------------------------------------------------------------------- Описание проекта: Цель проекта - решение одной из самых больших проблем в молекулярной биологии - вычисление 3-х мерной структуры белков из их аминокислотных последовательностей. Благодаря недавно завершенному проекту "Геном человека" известны аминокислотные последовательности всех белков в человеческом организме. Исследования по данному проекту также помогут в проектировании новых, не существующих белков. В случае успешного решения данных проблем мы сможем бороться с такими болезнями как рак, малярия, болезнь Альцгеймера, сибирская язва и другими генетическими и вирусными заболеваниями.[/quote] взято отсюда PROMO VIDEO new! Для большей научной пользы выставьте время рассчета ВЮ - 1 день. Для этого: 1. зайдите в Ваши настройки на сайте, http://boinc.bakerlab.org/rosetta/prefs.php?subset=project 2. выберите Target CPU run time = 1 day 3. нажмите Update 4. откройте БОИНК менеджер 5. если у вас сокращенный вид, нажмите Advanced View 6. выберите первую вкладку 7. выберите проект Розетта и нажмите кнопку "Обновить" сверху слева 8. Готово! Теперь каждая молекула будет "предсказываться" 1 день вместо 3 часов (по умолчанию) Статья в журнале LiveScience.com Мини FAQ Исследование болезней Что мы хотим знать о проекте Rosetta@Home ? Rosetta@Home FAQ Описание структур и методов моделирования белков Сравнение биомедицинских проектов распределенных вычислений Реферат по Росетте Инструкция по подключению на FreeBSD, боинк брать на оф сайте График ППД команды за последние 60 дней: (Show/Hide) Марка по теме проекта:  ---------------------------------------------------------------------------------------------------------- Статус сервера выдачи заданий:  Це повідомлення відредагував Rilian: May 10 2011, 13:12 |
  |
Відповідей
| gladiator_maximus |
 Apr 3 2020, 13:55 Apr 3 2020, 13:55
Пост
#2
|
 Все буде Україна!        Група: Moderators Повідомлень: 1 011 З нами з: 29-January 10 З: 47°52'N,35°03'E Користувач №: 1 284 Стать: Чол Free-DC_CPID Парк машин: ЦП-AMD FX 8300 3.6 GHz, ГП-Radeon RX 480 4 Gb; ОЗУ- DDR3 24 GB (1600MHz); SSD Iridium 256 Gb; HDD- Toshiba 2Tb+4Tb; / Intel® Xeon® CPU E5-2640 2.80GHz/16Gb DDR3 1333/RX 480 8 Gb/SSD 120 Gb |
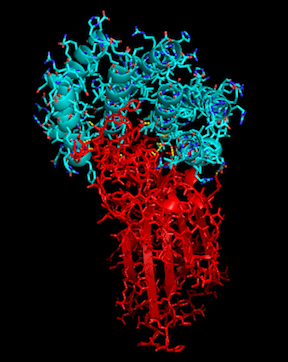 В связи с недавней вспышкой COVID-19 R@h использовался для прогнозирования структуры белков, важных для заболевания, а также для производства новых стабильных мини-белков, которые можно использовать в качестве потенциальной терапии и диагностики, таких как показанный выше, который связан с частью белка шипа COVID-19. Чтобы помочь нашим исследованиям, мы рады объявить о новом обновлении приложения, и благодаря помощи сообщества разработчиков Arm, включая Рекса Сент-Джона, Дмитрия Москальчука, Дэвида Тишлера, Ллойда Уоттса и Сахаджарупа, мы рады также включить платформу Linux-ARM. В этом обновлении мы продолжим делать белковые связующие для COVID-19 и связанных с ними задания с использованием новейшего источника Rosetta. Спасибо волонтерам R@h за постоянную поддержку этого проекта. Часы вашего процессора используются не только для точного моделирования структур важных белков, но и для разработки новых. Давайте объединимся и сразимся с COVID-19! 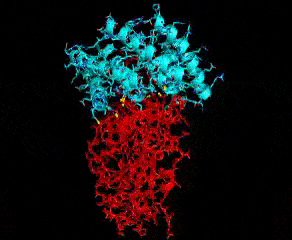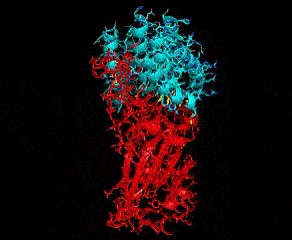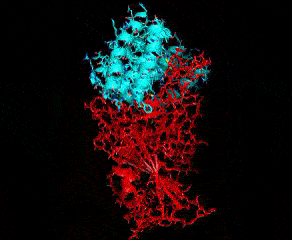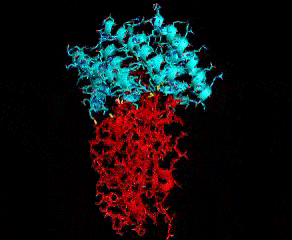 Основным способом взаимодействия белков друг с другом является прилипание друг к другу. Как вы могли видеть из графического приложения R@h, белки бывают разных форм и размеров. По этой причине большинство белков не прилипают случайным образом друг к другу, а скорее прилипают очень специфично к горстке других белков. Например, вирусный спайковый белок COVID-19 прилипает к человеческому белку ACE2, благодаря которому вирус попадает в клетку. IPD усердно работает над улучшением способности создавать такие связывающие взаимодействия. Этот процесс начинается с создания набора каркасных белков, которые не имеют иной цели, кроме точного сворачивания в атомную структуру. Эти строительные леса затем закрепляются на целевом белке, представляющем интерес, и их поверхности предназначены для идеального дополнения цели. Наконец, проекты оцениваются, фильтруются и тестируются на связывание в лаборатории. Теперь мы будем использовать R@h, чтобы выполнить этап проектирования поверхности. Стыковка и фильтрация выполняются быстро, но на самом деле разработка белка происходит медленно. Мы будем использовать огромное количество вычислительной мощности, доступной на R@h, для выборки каждой аминокислоты в каждой позиции на границе раздела. Затем мы выберем лучшие комбинации аминокислот с использованием имитации отжига и Монте-Карло. Выборка является ключевой для этого процесса, и поэтому мы обращаемся к R@h. Итак, присоединяйтесь к нам в ближайшие недели, когда мы создадим связующие вещества для COVID-19 и родственных белков. Мы все еще будем делать предсказание структуры и проектирование скаффолдов, поскольку они также крайне важны для белковой науки. Но обратите внимание на случаи разработки интерфейса, потому что кто-то может разрабатывать следующее лекарство от COVID-19. И, надеюсь, вы останетесь, как только пандемия закончится. Мы можем создавать такие связующие только потому, что много лет работали над этой проблемой. Впрочем, впереди еще долгий путь. Совершенствование науки требует времени и вычислений, поэтому мы надеемся, что вы присоединитесь к нам в этой увлекательной поездке. - Брайан Ковентри --------------------  |
Повідомлення у даній Темі
 Dimych Rosetta@home May 29 2007, 12:12
Dimych Rosetta@home May 29 2007, 12:12
 Arbalet Затея, на самом деле, очень хорошая и правильная... May 29 2007, 12:31
Arbalet Затея, на самом деле, очень хорошая и правильная... May 29 2007, 12:31
 Panda Хмм... Чи то я старий, чи "новое поколение вы... May 29 2007, 15:18
Panda Хмм... Чи то я старий, чи "новое поколение вы... May 29 2007, 15:18
 Rilian Если что мы напишем админам статистики или регистр... May 29 2007, 21:37
Rilian Если что мы напишем админам статистики или регистр... May 29 2007, 21:37

 nikelong
Если что мы напишем админам статистики или регист... Jun 1 2007, 06:09
nikelong
Если что мы напишем админам статистики или регист... Jun 1 2007, 06:09
 Rilian как админу команды могут и прислать. Jun 1 2007, 15:17
Rilian как админу команды могут и прислать. Jun 1 2007, 15:17
 Rilian Подключился к проекту. Посчитаю несколько дней Jun 16 2007, 14:04
Rilian Подключился к проекту. Посчитаю несколько дней Jun 16 2007, 14:04
 uNiUs Да, распылятся, пожалуй, не стоит. Если есть желан... Jun 17 2007, 23:17
uNiUs Да, распылятся, пожалуй, не стоит. Если есть желан... Jun 17 2007, 23:17
 Rilian так как ты капитан команды, довольно большой, я сч... Jun 17 2007, 23:27
Rilian так как ты капитан команды, довольно большой, я сч... Jun 17 2007, 23:27

 uNiUs
так как ты капитан команды, довольно большой, я с... Jun 18 2007, 00:08
uNiUs
так как ты капитан команды, довольно большой, я с... Jun 18 2007, 00:08
 Vladus
Полностью поддерживаю эту идею!
Призываю вс... Jun 18 2007, 01:20
Vladus
Полностью поддерживаю эту идею!
Призываю вс... Jun 18 2007, 01:20
 nikelong А через БОИНК-профиля некоторых людей можно-ж выло... Jun 18 2007, 06:31
nikelong А через БОИНК-профиля некоторых людей можно-ж выло... Jun 18 2007, 06:31

 uNiUs
А через БОИНК-профиля некоторых людей можно-ж выл... Jun 18 2007, 22:25
uNiUs
А через БОИНК-профиля некоторых людей можно-ж выл... Jun 18 2007, 22:25

 Vladus
А еще было бы неплохо не только объединить украин... Jun 19 2007, 01:36
Vladus
А еще было бы неплохо не только объединить украин... Jun 19 2007, 01:36

 uNiUs
Браво! Целое досье завел!
Жаль только, ч... Jun 19 2007, 09:13
uNiUs
Браво! Целое досье завел!
Жаль только, ч... Jun 19 2007, 09:13
 YuRi И через них выйти на капитанов их команд ;) Jun 18 2007, 13:35
YuRi И через них выйти на капитанов их команд ;) Jun 18 2007, 13:35
 ReMMeR погуглить по нику попытатся можно. Jun 18 2007, 23:05
ReMMeR погуглить по нику попытатся можно. Jun 18 2007, 23:05
 Oleg82 а чувак и недогадывается что гдето на него папочку... Jun 19 2007, 05:13
Oleg82 а чувак и недогадывается что гдето на него папочку... Jun 19 2007, 05:13

 Vladus
а чувак и недогадывается что гдето на нето папочк... Jun 19 2007, 09:36
Vladus
а чувак и недогадывается что гдето на нето папочк... Jun 19 2007, 09:36
 Rilian Что означают красные точки на графике?
http://i19... Jun 22 2007, 08:07
Rilian Что означают красные точки на графике?
http://i19... Jun 22 2007, 08:07  |
1 Користувачів переглядають дану тему (1 Гостей і 0 Прихованих Користувачів)
0 Користувачів:

|
Lo-Fi Версія | Поточний час: 14th December 2025 - 15:25 |












