Привіт Гість ( Вхід | Реєстрація )
| Dimych |
 May 29 2007, 12:12 May 29 2007, 12:12
Пост
#1
|
|
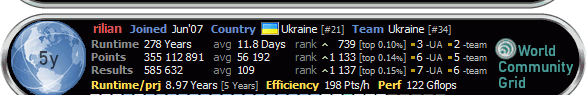
Так, я створив профіль! Група: New Members Повідомлень: 2 З нами з: 29-May 07 Користувач №: 522 Стать: Чол |
 Проект "Rosetta@home" ---------------------------------------------------------------------------------------------------------- Официальный сайт Официальная статистика по команде "Ukraine" ТОП-20 участников:  ---------------------------------------------------------------------------------------------------------- Дата основания команды - 12.04.2006 Капитан - uNiUs ---------------------------------------------------------------------------------------------------------- Для присоединения к команде Украины: 1. Загрузите BOINC менеджер (Если его у Вас еще нет!) 2. Перейдите в "расширенный вид" 3. Выберите сервис ---> добавить проект 4. Введите адрес проекта http://boinc.bakerlab.org/rosetta 5. Введите свои регистрационные данные. 6. Найдите нашу команду. Она называется Ukraine и адрес ее статистики вы могли видеть выше. 7. Если есть доступные для загрузки задания Вы их получите и начнете расчеты. ---------------------------------------------------------------------------------------------------------- BOINC 6 Versions Change Log ---------------------------------------------------------------------------------------------------------- Полезная информация: Для идентификации пользователя в BOINC могут служить 2 вещи: 1) пара e-mail/пароль 2) межпроектный идентификационный ID (Cross-project ID) - 32значное шестнадцатиричное число. Если Вы пожелаете подключится ещё и к другому BOINC-проекту, то помните: чтобы не плодить новых аккаунтов при подключении к новому проекту или команде, нужно обязательно везде регистрироваться с одним и тем же e-mail/паролем либо CPID. если при регистрации в проекте указать другие e-mail или пароль, BOINC создаст новый аккаунт с тем же именем! ---------------------------------------------------------------------------------------------------------- Если у вас есть желание помочь команде, посчитать на командный аккаунт, можно использовать так называемый "слабый ключ аккаунта" (weak account key) который позволяет коннектиться к проекту не вводя пароли. Все очки будут присваиваться командному юзеру distributed.org.ua Чтобы подключиться: 0) установите BOINC 1) создайте файл account_boinc.bakerlab.org_rosetta.xml в папке \data\ 2) в файл поместите текст CODE <account> <master_url>http://boinc.bakerlab.org/rosetta/</master_url> <authenticator>46ed7286c5974c66fe43ee6b4828413a</authenticator> <project_name>rosetta@home</project_name> </account> 3) перезагрузите BOINC ---------------------------------------------------------------------------------------------------------- Описание проекта: Цель проекта - решение одной из самых больших проблем в молекулярной биологии - вычисление 3-х мерной структуры белков из их аминокислотных последовательностей. Благодаря недавно завершенному проекту "Геном человека" известны аминокислотные последовательности всех белков в человеческом организме. Исследования по данному проекту также помогут в проектировании новых, не существующих белков. В случае успешного решения данных проблем мы сможем бороться с такими болезнями как рак, малярия, болезнь Альцгеймера, сибирская язва и другими генетическими и вирусными заболеваниями.[/quote] взято отсюда PROMO VIDEO new! Для большей научной пользы выставьте время рассчета ВЮ - 1 день. Для этого: 1. зайдите в Ваши настройки на сайте, http://boinc.bakerlab.org/rosetta/prefs.php?subset=project 2. выберите Target CPU run time = 1 day 3. нажмите Update 4. откройте БОИНК менеджер 5. если у вас сокращенный вид, нажмите Advanced View 6. выберите первую вкладку 7. выберите проект Розетта и нажмите кнопку "Обновить" сверху слева 8. Готово! Теперь каждая молекула будет "предсказываться" 1 день вместо 3 часов (по умолчанию) Статья в журнале LiveScience.com Мини FAQ Исследование болезней Что мы хотим знать о проекте Rosetta@Home ? Rosetta@Home FAQ Описание структур и методов моделирования белков Сравнение биомедицинских проектов распределенных вычислений Реферат по Росетте Инструкция по подключению на FreeBSD, боинк брать на оф сайте График ППД команды за последние 60 дней: (Show/Hide) Марка по теме проекта:  ---------------------------------------------------------------------------------------------------------- Статус сервера выдачи заданий:  Це повідомлення відредагував Rilian: May 10 2011, 13:12 |
  |
Відповідей
| Rilian |
 Jun 24 2013, 14:28 Jun 24 2013, 14:28
Пост
#2
|
 interstellar           Група: Team member Повідомлень: 17 171 З нами з: 22-February 06 З: Торонто Користувач №: 184 Стать: НеСкажу Free-DC_CPID Парк машин: 2 ноути і 4 компа |
To our awesome Rosetta@Home contributors:
At the beginning of 2012 we told you that it looked like eight designed proteins stuck to EED as desired. Further experiments showed us that of our eight initial hits, only two were real, and those two proteins were too weak to be improved with laboratory techniques. We've gone back to the drawing board, using more aggressive modeling techniques to design better binding proteins to mimic Ezh2. It turns out that no known protein structure is similar enough to the Ezh2 binding helix to allow us to simply transplant key amino acids from Ezh2 to a new host protein, as we tried before. What were doing now is making new proteins from scratch that have the exact shape that we need to mimic Ezh2. The "EED" runs you'll now see on Rosetta@Home are structure prediction of designs to see if they have the desired curvature when Rosetta folds them up. Thanks again for all of your donated computer time! For those new to this thread/message board, here is what I wrote previously about the EED project: The second of these interactions involve a protein called EED and another called Ezh2. If you think about DNA as a ladder, then each of our cells has about six billion rungs worth of ladder. In order to keep all of that DNA from getting hopelessly tangled, our cells keep it on little spools, called histones, when not in use. Think of this like a massive film archive. It turns out that there is a lot of DNA that never gets used, so the cells put special flags on those spools (histones) that say something like "never use this section of DNA". EED is a large protein that attaches to those flags and brings along Ezh2, a "flagging machine" for the ride. EED makes sure that Ezh2 adds flags to histones that are supposed to get them, and not to histones carrying DNA the cell actually wants to use. Some cancer cells reduce the amounts of EED or Ezh2 to prevent flagging of DNA regions that they want to use to take over our bodies. Other cancer cells expand the amounts of EED or Ezh2 to flag DNA regions that are getting in the way. Scientists are working hard to better understand how EED, Ezh2, and friends work in normal cells and what goes wrong in cancer cells. Some are trying to develop drugs that prevent Ezh2 from attaching to EED. This means that the drugs have to stick to EED more tightly than Ezh2. Although Ezh2 attaches relatively loosely to a large patch on the surface of EED, their aren't any drugs yet available that do what we want. I'm trying to make proteins that will do the same job. I used Rosetta@home to design a set of proteins that mimic Ezh2 in order to block it from attaching to EED. Just to give you a sense of how much computing I need for a project like this, I submitted just under two million work units to Rosetta@home, and donor's computers ran my protocol just under a billion times to give me about 54 designs to look at, from which fourteen were suitable for testing. This took about a month to run on Rosetta@home. From initial experiments, it looks like eight of the designs stick to EED, and one in particular sticks three times better than the Ezh2 found in our cells. Now I'm working to improve the best design at the lab bench and hope to send it to co-workers for testing in living cells. There's still a lot of work to be done to make sure that everything is working right with this design, but I want to give a big thank-you to all of you who donated your computer time to make this possible! For more information about EED, Ezh2, and related proteins, please visit: http://en.wikipedia.org/wiki/EED http://en.wikipedia.org/wiki/SUZ12 http://en.wikipedia.org/wiki/EZH2 http://en.wikipedia.org/wiki/PRC2 http://en.wikipedia.org/wiki/Polycomb-group_proteins Work units for this project carried the word "EED" in their name. -------------------- |
Повідомлення у даній Темі
 Dimych Rosetta@home May 29 2007, 12:12
Dimych Rosetta@home May 29 2007, 12:12
 Arbalet Затея, на самом деле, очень хорошая и правильная... May 29 2007, 12:31
Arbalet Затея, на самом деле, очень хорошая и правильная... May 29 2007, 12:31
 Panda Хмм... Чи то я старий, чи "новое поколение вы... May 29 2007, 15:18
Panda Хмм... Чи то я старий, чи "новое поколение вы... May 29 2007, 15:18
 Rilian Если что мы напишем админам статистики или регистр... May 29 2007, 21:37
Rilian Если что мы напишем админам статистики или регистр... May 29 2007, 21:37

 nikelong
Если что мы напишем админам статистики или регист... Jun 1 2007, 06:09
nikelong
Если что мы напишем админам статистики или регист... Jun 1 2007, 06:09
 Rilian как админу команды могут и прислать. Jun 1 2007, 15:17
Rilian как админу команды могут и прислать. Jun 1 2007, 15:17
 Rilian Подключился к проекту. Посчитаю несколько дней Jun 16 2007, 14:04
Rilian Подключился к проекту. Посчитаю несколько дней Jun 16 2007, 14:04
 uNiUs Да, распылятся, пожалуй, не стоит. Если есть желан... Jun 17 2007, 23:17
uNiUs Да, распылятся, пожалуй, не стоит. Если есть желан... Jun 17 2007, 23:17
 Rilian так как ты капитан команды, довольно большой, я сч... Jun 17 2007, 23:27
Rilian так как ты капитан команды, довольно большой, я сч... Jun 17 2007, 23:27

 uNiUs
так как ты капитан команды, довольно большой, я с... Jun 18 2007, 00:08
uNiUs
так как ты капитан команды, довольно большой, я с... Jun 18 2007, 00:08
 Vladus
Полностью поддерживаю эту идею!
Призываю вс... Jun 18 2007, 01:20
Vladus
Полностью поддерживаю эту идею!
Призываю вс... Jun 18 2007, 01:20
 nikelong А через БОИНК-профиля некоторых людей можно-ж выло... Jun 18 2007, 06:31
nikelong А через БОИНК-профиля некоторых людей можно-ж выло... Jun 18 2007, 06:31

 uNiUs
А через БОИНК-профиля некоторых людей можно-ж выл... Jun 18 2007, 22:25
uNiUs
А через БОИНК-профиля некоторых людей можно-ж выл... Jun 18 2007, 22:25

 Vladus
А еще было бы неплохо не только объединить украин... Jun 19 2007, 01:36
Vladus
А еще было бы неплохо не только объединить украин... Jun 19 2007, 01:36

 uNiUs
Браво! Целое досье завел!
Жаль только, ч... Jun 19 2007, 09:13
uNiUs
Браво! Целое досье завел!
Жаль только, ч... Jun 19 2007, 09:13
 YuRi И через них выйти на капитанов их команд ;) Jun 18 2007, 13:35
YuRi И через них выйти на капитанов их команд ;) Jun 18 2007, 13:35
 ReMMeR погуглить по нику попытатся можно. Jun 18 2007, 23:05
ReMMeR погуглить по нику попытатся можно. Jun 18 2007, 23:05
 Oleg82 а чувак и недогадывается что гдето на него папочку... Jun 19 2007, 05:13
Oleg82 а чувак и недогадывается что гдето на него папочку... Jun 19 2007, 05:13

 Vladus
а чувак и недогадывается что гдето на нето папочк... Jun 19 2007, 09:36
Vladus
а чувак и недогадывается что гдето на нето папочк... Jun 19 2007, 09:36
 Rilian Что означают красные точки на графике?
http://i19... Jun 22 2007, 08:07
Rilian Что означают красные точки на графике?
http://i19... Jun 22 2007, 08:07  |
2 Користувачів переглядають дану тему (2 Гостей і 0 Прихованих Користувачів)
0 Користувачів:

|
Lo-Fi Версія | Поточний час: 3rd February 2026 - 17:12 |