Привіт Гість ( Вхід | Реєстрація )
| Rilian |
 Dec 21 2008, 04:23 Dec 21 2008, 04:23
Пост
#1
|
 interstellar           Група: Team member Повідомлень: 17 160 З нами з: 22-February 06 З: Торонто Користувач №: 184 Стать: НеСкажу Free-DC_CPID Парк машин: ноут и кусок сервера |
 Помоги вылечить мускульную дистрофию, 2 фаза / Help Cure Muscular Dystrophy, Phase 2 Как присоединиться читайте в главном топике World Community Grid
 Введение В 1986 году был идентифицирован первый ген (dystrophin gene), вовлеченный в дистрофию Дюшена, самую общую форму мышечной дистрофии. Благодаря генетическим исследованиям сегодня известно более 200 генов, вызывающих нервно-мышечные болезни. Однако, до сих пор знания о функциях и взаимодействии между собой белков, закодированных этими генами, остаются крайне незначительными. Раскрытие механизмов возникновения и развития нервно-мышечных заболеваний (патогенез), учитывая большое количество вовлеченных в него генов и кодируемых ими белков, становится все более сложной задачей по мере увеличения наших знаний в этой области. Все клетки содержат одинаковую генетическую информацию в виде ДНК, которая в целом образует геном организма. Процесс экспрессии генов (у человека найдено более 20 тысяч генов), содержащихся в геноме, приводит к образованию белков (более 45 тысяч, т.е. в среднем один ген кодирует 1,6 белков), без которых невозможно функционирование клеток, в том числе специфических, например, мышечных. Некоторые их этих белков являются ферментами, другие - сигнальными молекулами, а могут быть и рецепторами, специфически связывающиеся с другими молекулами (лигандами); а также - структурными белками. Конформация (геометрическая форма, которую принимает органическая молекула) белка в пространстве (3D-структура) определяет его природные функции. Функционирование мышц зависит от многих белков. Эти белки локализованы и работают на различных уровнях - в оболочке клетки, ее протопласте (внутреннем содержимом) или в аксоне двигательного нерва, который соединяет нерв с мышечной тканью и передает команды нервной системы. Большинство нервно-мышечных болезней возникает под действием генетических изменений (мутации в определенных генах вызывают изменения в трехмерной структуре кодируемого ими белка), приводящих к утрате белками полностью или частично их первоначальных функций или к прекращению синтезирования белка как такового. Нервно-мышечные заболевания можно классифицировать по белкам или участкам белков, видоизменившихся под действием мутации генов. Поэтому, чтобы разобраться в природе того или иного нервно-мышечного заболевания, исследователи стремятся связать их с нарушением функциональности определенных белков. Необходимо более точное понимание взаимодействий и функций белков, вовлеченных в патогенез той или иной мышечной дистрофии. Невозможно переоценить значимость данной информации в разработке терапевтических процедур и инновационных методов лечения большого количества нервно-мышечных заболеваний. Цель проекта В Help Cure Muscular Dystrophy прибегают к помощи World Community Grid в определении белок-белковых взаимодействий более 2 200 безызбыточных белков с известной структурой, информация о которых содержится в Белковом Банке данных (www.rcsb.org/pdb). Исследоваться будут также белки, которые синтезируются в результате экспрессии мутировавших генов. Целью проекта является создание новой базы данных с информацией о функционально взаимодействующих белках. Дальнейшие исследования будут связаны с изучением участков белков, вовлеченных во взаимодействия лигандов (например, лекарств) с ДНК. Эта тема представляет значительный медицинский интерес, хотя сейчас упор делается на проектировании малых молекул (лигандов), которые нарушают (ингибируют) или улучшают работу определенных белков, намного сложнее определить, как та же самая малая молекула может прямо или косвенно влиять на другие взаимодействия белков. Исследовательский подход В этом проекте используется подход, в котором скомбинирована информация об эволюции (как развитие изменило белки и определило их функции) и молекулярное моделирование (вычислительное определение относительного положения двух взаимодействующих белковых партнеров), чтобы идентифицировать потенциальные взаимодействия. Молекулярное моделирование объединяет теоретические методы и вычислительные способы для моделирования поведения биологических молекул. Эти методы и способы применяются для исследования структуры биологических систем, таких как свернувшиеся белки, или при идентификации белков-лигандов, связывающихся с небольшими химическими системами, крупными биомолекулами и белковыми комплексами. Докинг (стыковка) белок-лиганд - молекулярная техника моделирования, которая прогнозирует положение и ориентацию (трехмерную структуру) белка относительно лиганда (который может быть другим белком, ДНК или лекарственным средством). Методы молекулярного докинга основаны исключительно на физических принципах — даже белки с неизвестными или слабо изученными функциями могут быть исследованы на предмет стыковки с различными лигандами. Единственным обязательным условием является знание трехмерной структуры белков, полученное теоретическими методами или в ходе экспериментального исследования. При использовании молекулярного докинга производится перебор известных молекул из баз данных с целью поиска вариантов, обнаруживающих сродство, т.е. хорошо связывающихся друг с другом. Степень сродства определяется с помощью оценочной функции по геометрическим и химическим параметрам полученного соединения. Геометрия оценивается по тому, насколько хорошо сочетаются трехмерные структуры белка и лиганда (должны быть подогнаны как рука под перчатку). С химической точки зрения оценивается сила межатомных взаимодействий между белком и лигандом. Для таких сложных структур как белки (наименьшие из которых содержат сотни атомов) исследования белок-белковых взаимодействий могут потребовать значительных вычислительных ресурсов. Без World Community Grid вычисления, необходимые для моделирования молекулярного докинга, были бы слишком трудоёмкими. Для первых 168 белков, изучавшихся еще в первой фазе проекта, привлеченное с помощью WCG процессорное время составило 8 000 лет. Во второй фазе проекта, где будут исследоваться уже 2 246 белков, предполагаемое время расчетов достигнет 91 680 лет. Преодолеть этот вычислительный барьер поможет информация об эволюции белков, позволяющая предсказывать взаимодействующие участки белков. Этот предварительный анализ уменьшает предполагаемое время вычислений в 100 раз, тем самым увеличивая количество исследуемых белков. Тем не менее, без помощи World Community Grid запланированные вычисления окажутся невыполнимыми. Добровольцы, жертвующие свободные вычислительные ресурсы World Community Grid, высвободят мощности для других исследований, проводимых AFM, CNRS, INSERM и пр. научными игроками, направленных на развитие научных инструментов, которые увеличивают наши знания о природе и методах лечения редких заболеваний. Исследователи Вычислительная биология: * Dr. Alessandra Carbone, Analytical Genomics, FRE3214 CNRS-UPMC, Universitй Pierre et Marie Curie, Paris. * Yann Ponty, Department of Computer Science, Universitй Pierre et Marie Curie, Paris. ГРИД-технологии: * Jean-Marie Chesneaux team, Computer Science Laboratory of Paris 6 (LIP6), CNRS Laboratory UMR 7606, Universitй Pierre et Marie Curie, Paris. Миопатия (заболевание мышц): * Pascale Guicheney team, INSERM Laboratory U582, Myology Institut, "Pitiй-Salpetriиre" Hospital, Paris. Молекулярное моделирование: * Richard Lavery, Institut de Biologie et Chimie des Protйines, CNRS UMR 5086, Universitй de Lyon. * Sophie Sacquin-Mora, Laboratory of theoretical biochemistry, CNRS Laboratory UPR9080, "Institut de Biologie Physico-Chimique", Paris. Вопросы и ответы по проекту: (Show/Hide) Скриншот графического клиента  График производительности проекта:  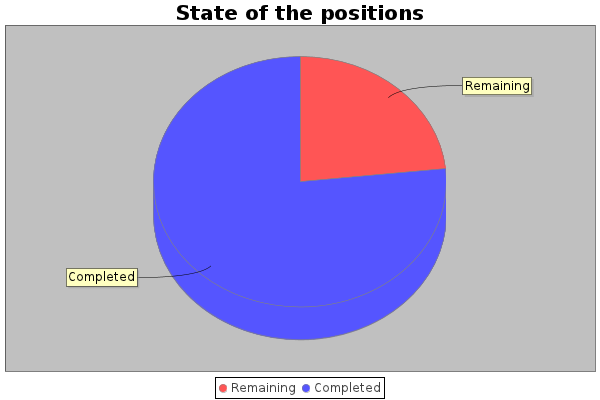 Це повідомлення відредагував Rilian: Dec 11 2010, 14:10 |
  |
Відповідей(105 - 119)
| Rilian |
 Jul 21 2012, 20:04 Jul 21 2012, 20:04
Пост
#106
|
 interstellar           Група: Team member Повідомлень: 17 160 З нами з: 22-February 06 З: Торонто Користувач №: 184 Стать: НеСкажу Free-DC_CPID Парк машин: ноут и кусок сервера |
Стат на офсайте уже год не обновлялся
-------------------- |
| Death |
 Jul 21 2012, 21:47 Jul 21 2012, 21:47
Пост
#107
|
 <script ///>           Група: Moderators Повідомлень: 6 371 З нами з: 5-November 03 З: Kyiv Користувач №: 26 Стать: НеСкажу Free-DC_CPID Парк машин: гидропарк jabber:deadjdona@gmail.com |
хотел сказать "ну да это же вцг", но потом передумал
-------------------- |
| re_SET |
 Jul 25 2012, 10:06 Jul 25 2012, 10:06
Пост
#108
|
 кранчер зі стажем       Група: Trusted Members Повідомлень: 455 З нами з: 26-January 09 З: Волинська обл., смт. Любешів Користувач №: 911 Стать: Чол Парк машин: 1х Athlon X2 BE2300 (2.2 Ghz) 1x C2D E8200 1x C2D E2180 (2.34 Ghz) 1x C2D E2160 1x C2D E5200 (2.9 Ghz) 9x C2D E6500 (Up 3.24 Ghz) 1x C2Q Q8300 (Up 3 Ghz) |
Пишут что досчитается через 11 дней Задания еще есть! Вопреки побаиваниям успеваю накранчить два года в проекте )) -------------------- Нельзя попасть в Рай одной религии, не попав при этом в Ад всех остальных...
           (Show/Hide) |
| Bel |
 Aug 11 2012, 08:47 Aug 11 2012, 08:47
Пост
#109
|
 Мега ранчер         Група: Trusted Members Повідомлень: 1 287 З нами з: 3-September 10 Користувач №: 1 476 Стать: Чол |
"I know our prior estimates for the completion date indicated that we would be done already. However, those estimates did not factor in delays required to handle the children and grand-children work units. Our new estimate is that we will complete this project in a little more than 5 weeks from now. We expect the researcher to provide an update soon after that. Thanks go to everyone who has contributed to this project."
|
| Rilian |
 Aug 19 2012, 22:26 Aug 19 2012, 22:26
Пост
#110
|
 interstellar           Група: Team member Повідомлень: 17 160 З нами з: 22-February 06 З: Торонто Користувач №: 184 Стать: НеСкажу Free-DC_CPID Парк машин: ноут и кусок сервера |
Here are some (hopefully accurate) information summarizing some scientist posts sent during the last 3 years.
Because of the computation complexity, the screening has be planned to be performed in several steps. HCMD1 served to realize a first screening for identifying candidates for more detailed investigation and for identifying the most appropriate docking areas. Based on HCMD1 results, HCMD2 served to investigate more deeply the identified candidates. The current "re-work" (since Spring 2012) served to challenge some of HCMD2-results. Maybe, many of the members are not really aware about pharmaceutical and medical research. In all cases, what we are doing at WCG is real research life: Planning Investigating Challenging Sorting (interesting/promising vs. not/less interesting/promising) Planning the next step Investigating Challenging Sorting ... For one final pharmaceutical product, it is usually necessary to investigate recursively at least 10'000 substances. This "success rate" is also similar for agro-business products (e.g. crop protection). Worldwide, many scientists will work a life long on problems without experiencing a concrete and full success. In vitro and in silico (computational bio-science) researches are necessary for helping to identify "faster" the "right" product. Pharma research and development require for a new product at least between 7 and 10 years; some products require much more. In 2008, Raphaël Bolze wrote his PhD thesis about the HCMD project. It is a very interesting and explaining document about the work we support with HCMD. Enjoy, Yves -------------------- |
| Rilian |
 Aug 22 2012, 10:08 Aug 22 2012, 10:08
Пост
#111
|
 interstellar           Група: Team member Повідомлень: 17 160 З нами з: 22-February 06 З: Торонто Користувач №: 184 Стать: НеСкажу Free-DC_CPID Парк машин: ноут и кусок сервера |
THANK YOU from all the scientists of the HCMD project
Dear All, We are at the end of HCMD2 and I would like to thank you for the patience and persistence in running our docking program on your machines. The huge amount of cross-docking data that we collected, thanks to you (!), has been realized for the first time. It is a mine of information for our research in protein-protein interactions and it will also constitute a precious amount of information for our colleagues around the world interested in molecular docking. We finished analyzing the data on the 168 protein complexes run on HCMD1 and we now know what has to be done next. We shall integrate novel and quantitative, experimental data on protein binding to predict not only the conformation of interacting proteins, but also which proteins will interact and how strongly. This involves four specific challenges: 1) Obtain quantitative experimental data on protein interactions with a wide range of binding affinities. We will use surface plasmon resonance (SPR), followed by isothermal titration calorimetry (ITC) to fully characterize the thermodynamics of protein interactions over a wide range of affinities and physical conditions (concentration, pH, temperature, …). These methods constitute ideal tools for our purpose. They will be used to quantify interactions between a set of commercially available proteins, including known interacting partners. However, we will also characterize nominally non-functional "cross-interactions" within this set to test, for the first time, the common assumption that choosing single proteins from known binary complexes, or choosing proteins from different cellular compartments, implies the absence of interaction. 2) Use evolutionary sequence data to detect protein residues involved in interaction interfaces and pairs of interacting proteins. We will identify key residues within interaction sites and co-evolution signals between pairs of interaction sites in order to predict interacting partners and integrate this information into a refined molecular docking approach, with the aim of identifying binary interactions within a large set of proteins. This goal will include constructing an automated pipeline for co-evolution analysis of single proteins and protein pairs. 3) Formulate new protein-protein interaction potentials using experimental data, molecular simulations and existing structural data. Molecular simulations coupled with free energy calculations will be used to obtain an atomic-scale view of the dissociation of a limited number of the weak and strong protein interactions studied by microcalorimetry. We will determine the extent to which complexes have well-defined conformations and fully desolvated interfaces. This data will be used to formulate and iteratively refine new interaction potentials within a coarse-grain model, which will be sensitive to binding affinity. 4) Carry out a refined analysis of the large database of protein interactions that you generated to characterize interaction networks and binding promiscuity. During stage two of the Help Cure Muscular Dystrophy project (HCMD2), the resources of World Community Grid were used to dock all possible protein pairs within a set of 2200 proteins, potentially important for understanding and treating neurodegenerative diseases. This data will be analyzed to characterize key “hub” proteins and network structures, first, with the existing energetic and residue conservation data and then with the new methods resulting from 1-3. The methods and interaction data derived from our studies will be freely available to the scientific community by the implementation of web servers and web databases. We will do all this with 4 years funding from the French ministry of research that was awarded to our group this year. We shall devote this grant to the development of the new tools (in biophysics and bioinformatics) mentioned above, as well as on the analysis of the HCMD2 dataset to arrive to the best prediction possible on the human protein-protein interaction network that you generated in these last two years. To keep you informed on the development of the project, I shall provide news on the advancements in my webpage. Pointers to the publications will be given there. If by any chance I do not post news from more than 6 months, send me a reminder! THANK YOU again to all of you from all the scientists of the HCMD1 and HCMD2 projects. Best regards, Alessandra -------------------- |
| Rilian |
 Aug 29 2012, 20:02 Aug 29 2012, 20:02
Пост
#112
|
 interstellar           Група: Team member Повідомлень: 17 160 З нами з: 22-February 06 З: Торонто Користувач №: 184 Стать: НеСкажу Free-DC_CPID Парк машин: ноут и кусок сервера |
еще 25 дней работы
QUOTE We have 355,685 'children' and 413,437 'grand children' workunits currently waiting to loaded and run. We also have 2 days worth of work currently loaded in that is waiting to run. We have been finishing about 40,000 workunits per day. That leaves us with about 20 days of work. Given that additional grand-children and some great grand children will be generated during the running. We would expect to see somewhere around 25 days of constant work available. -------------------- |
| Rilian |
 Sep 14 2012, 18:42 Sep 14 2012, 18:42
Пост
#113
|
 interstellar           Група: Team member Повідомлень: 17 160 З нами з: 22-February 06 З: Торонто Користувач №: 184 Стать: НеСкажу Free-DC_CPID Парк машин: ноут и кусок сервера |
QUOTE We have about 17 days of work to go based on the 500,000 grandchildren and estimated 50-75k great-grandchildren. -------------------- |
| Death |
 Sep 14 2012, 19:28 Sep 14 2012, 19:28
Пост
#114
|
 <script ///>           Група: Moderators Повідомлень: 6 371 З нами з: 5-November 03 З: Kyiv Користувач №: 26 Стать: НеСкажу Free-DC_CPID Парк машин: гидропарк jabber:deadjdona@gmail.com |
QUOTE We have about 17 days of work to go и мышечная дистрофия будет излечима? -------------------- |
| Rilian |
 Sep 15 2012, 07:24 Sep 15 2012, 07:24
Пост
#115
|
 interstellar           Група: Team member Повідомлень: 17 160 З нами з: 22-February 06 З: Торонто Користувач №: 184 Стать: НеСкажу Free-DC_CPID Парк машин: ноут и кусок сервера |
Там ведутся серьезные работы на эту тему. Читай выше, французы дали грант лаборатории 4 года изучать результаты полученные в этой фазе проекта
-------------------- |
| Rilian |
 Sep 21 2012, 14:43 Sep 21 2012, 14:43
Пост
#116
|
 interstellar           Група: Team member Повідомлень: 17 160 З нами з: 22-February 06 З: Торонто Користувач №: 184 Стать: НеСкажу Free-DC_CPID Парк машин: ноут и кусок сервера |
Тасков осталось на 9 дней!
-------------------- |
| Rilian |
 Sep 26 2012, 00:21 Sep 26 2012, 00:21
Пост
#117
|
 interstellar           Група: Team member Повідомлень: 17 160 З нами з: 22-February 06 З: Торонто Користувач №: 184 Стать: НеСкажу Free-DC_CPID Парк машин: ноут и кусок сервера |
По прогнозам осталось 5 дней до конца выдачи заданий!
http://i137.photobucket.com/albums/q210/Se...CGDashboard.png -------------------- |
| Rilian |
 Sep 28 2012, 10:47 Sep 28 2012, 10:47
Пост
#118
|
 interstellar           Група: Team member Повідомлень: 17 160 З нами з: 22-February 06 З: Торонто Користувач №: 184 Стать: НеСкажу Free-DC_CPID Парк машин: ноут и кусок сервера |
26 Sep 2012
End of the Help Cure Muscular Dystrophy - Phase 2 project Проект почти завершен, выдаются последние задания Summary As a result of the generous contribution of computing power from our members, the Help Cure Muscular Dystrophy - Phase 2 project is on the verge of finishing. World Community Grid is pleased to announce, that as a result of the generous contribution of computing power from our members, the Help Cure Muscular Dystrophy - Phase 2 project is very close to being completed. The Help Cure Muscular Dystrophy - Phase 2 project was launched on May 12, 2009. To date, World Community Grid members have processed over 113 million results which required nearly 53,000 years of computing power. This work would have taken too many years to even be attempted, using the computing resources available to the researchers at the Université Pierre et Marie Curie and joint research facilities. Using World Community Grid, this research was completed in less than 2.5 years. The researchers are now working on analyzing the results data to determine the more detailed protein-to-protein interactions involved with neuromuscular diseases. They expect to publish their results in public databases, along with descriptive papers. You may read about these plans in this forum post by Dr. Alessandra Carbone, the lead researcher on the Help Cure Muscular Dystrophy - Phase 2 project. If you contributed your computer power to the Help Cure Muscular Dystrophy - Phase 2 research project, the staff at the Université Pierre et Marie Curie in Paris wish to express their sincere gratitude to you. Don't forget - we still need your help with the other active research projects running on World Community Grid! All of these important projects need your computer time. If you had selected to only contribute to the Help Cure Muscular Dystrophy - Phase 2 project, please go to your My Projects page to review and update your project selection. все у кого был включен только этот проект, зайдите в профиль и выставьте еще проектов, чтобы компы не простаивали когда задания закончатся -------------------- |
| Rilian |
 Oct 3 2012, 11:56 Oct 3 2012, 11:56
Пост
#119
|
 interstellar           Група: Team member Повідомлень: 17 160 З нами з: 22-February 06 З: Торонто Користувач №: 184 Стать: НеСкажу Free-DC_CPID Парк машин: ноут и кусок сервера |
QUOTE Out of Steady Work We have finally reached the end of steady work for the project. Due to the continued generation of workunits, there will be intermittent work for sometime yet, but it will not be regularly available. Thank you all for getting us to this point!!!!!! http://www.worldcommunitygrid.org/forums/w...ad_thread,33916 -------------------- |
| Rilian |
 Oct 3 2012, 16:14 Oct 3 2012, 16:14
Пост
#120
|
 interstellar           Група: Team member Повідомлень: 17 160 З нами з: 22-February 06 З: Торонто Користувач №: 184 Стать: НеСкажу Free-DC_CPID Парк машин: ноут и кусок сервера |
Задания закончились
еще гдето 2 недели будут высылаться повторные но если у вас включен только этот проект рекомендую проверить настройки и добавить другие проекты -------------------- |
  |
2 Користувачів переглядають дану тему (2 Гостей і 0 Прихованих Користувачів)
0 Користувачів:

|
Lo-Fi Версія | Поточний час: 1st July 2025 - 12:55 |

















 Dead J
Dead J



